CRISPR(规律成簇间隔短回文序列)和与其功能相关的蛋白质(CRISPR-associated,Cas)组成的CRISPR-Cas系统是细菌和古细菌在不断进化的过程中获得的一种适应性免疫防御机制[1]。III型CRISPR系统区别于其它CRISPR系统的一个重要特征是拥有类似人体cGAS-STING天然免疫信号通路的cOA(cyclic oligoadenylate,环化寡腺苷酸)信号通路[2-3]。当外源RNA或DNA入侵时,该系统中的Cas10蛋白会被激活并合成cOA,随后cOA分子作为第二信使激活一系列的辅助核酸酶以促使后者对DNA或RNA进行非特异性地切割[2-3]。然而,cOA信号通路的过度的激活将导致宿主菌的休眠或死亡[4]。2018年,苏格兰圣安德鲁斯大学研究组首次鉴定出了一类能专一降解cOA的蛋白如Sso2081和Sso1393,并将其命名为CRISPR环状核酸酶(CRISPR ring nuclease, Crn)[5]。研究表明,Sso2081可将cA4(tetra-adenylate,环化四腺苷酸)切割为5ˊ-OH-ApA-2ˊ, 3ˊ-环磷酸(A2>P),从而关闭cA4信号通路5。然而,关于Sso2081识别和切割底物的分子机制一直不为人们所知。

该研究以X-射线晶体学为主要研究手段,解析了环核酸酶家族成员Sso2081及其底物、切割中间产物和最终产物等多个催化阶段的晶体结构(图1,图2)。Sso2081在整体结构上是一个形状酷似蝴蝶的同源二聚体,与其他环状核酸酶相比,其最突出的结构特征是C末端的Helical insert(HI)。在APO状态下,HI呈开放状态,以便于底物进入催化中心。当配体结合(磷酸根离子或cA4)时,HI转变为封闭状态从而将底物分子锁定在活性口袋中。因此,该结构模型揭示了Sso2081以一种门控机制(gate-locking mechanism)来识别和结合配体。
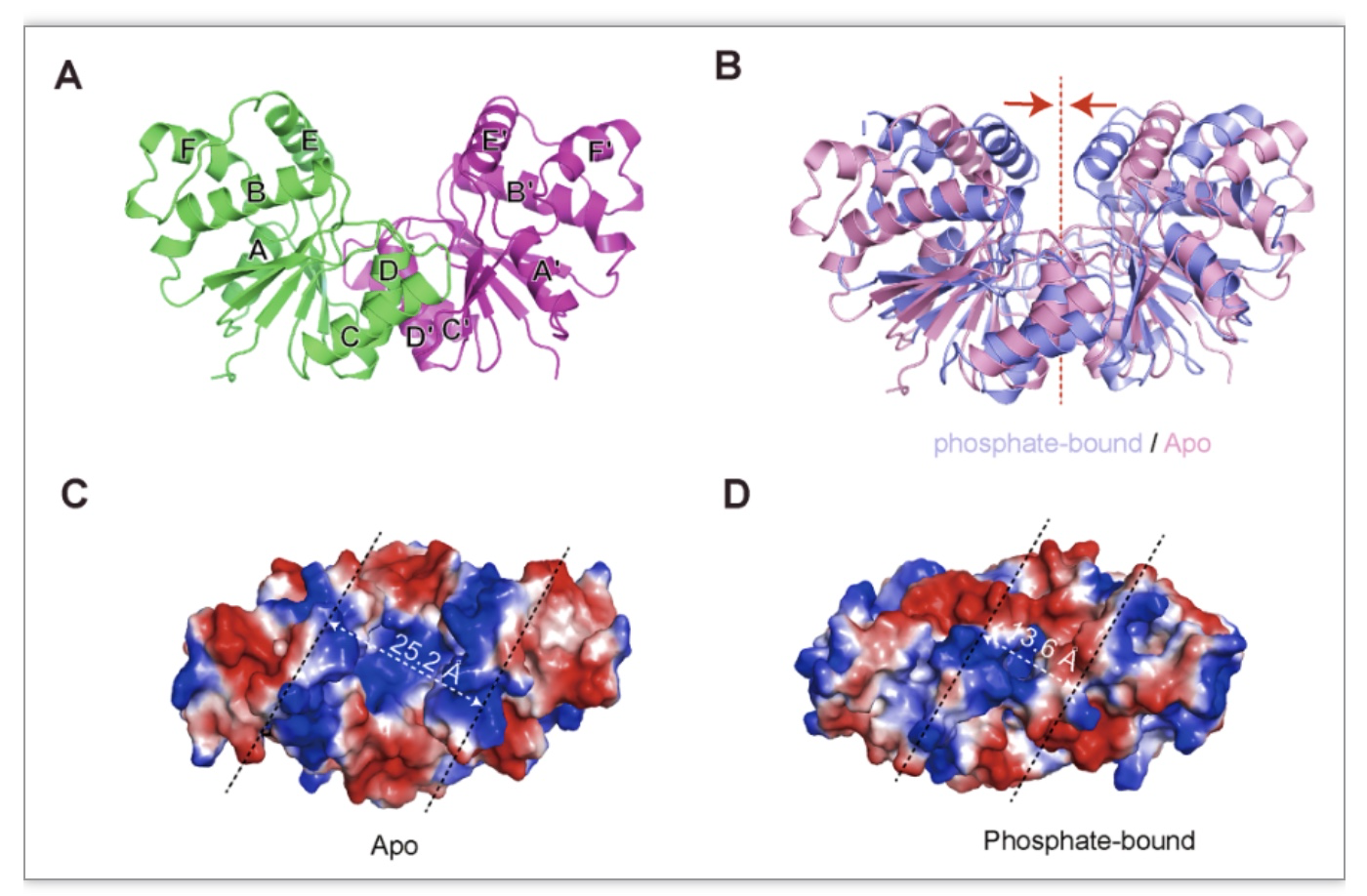
图1. Sso2081及其在磷酸根结合状态下的晶体结构
(图源:Du L , et al., Nucleic Acids Res. 2023)
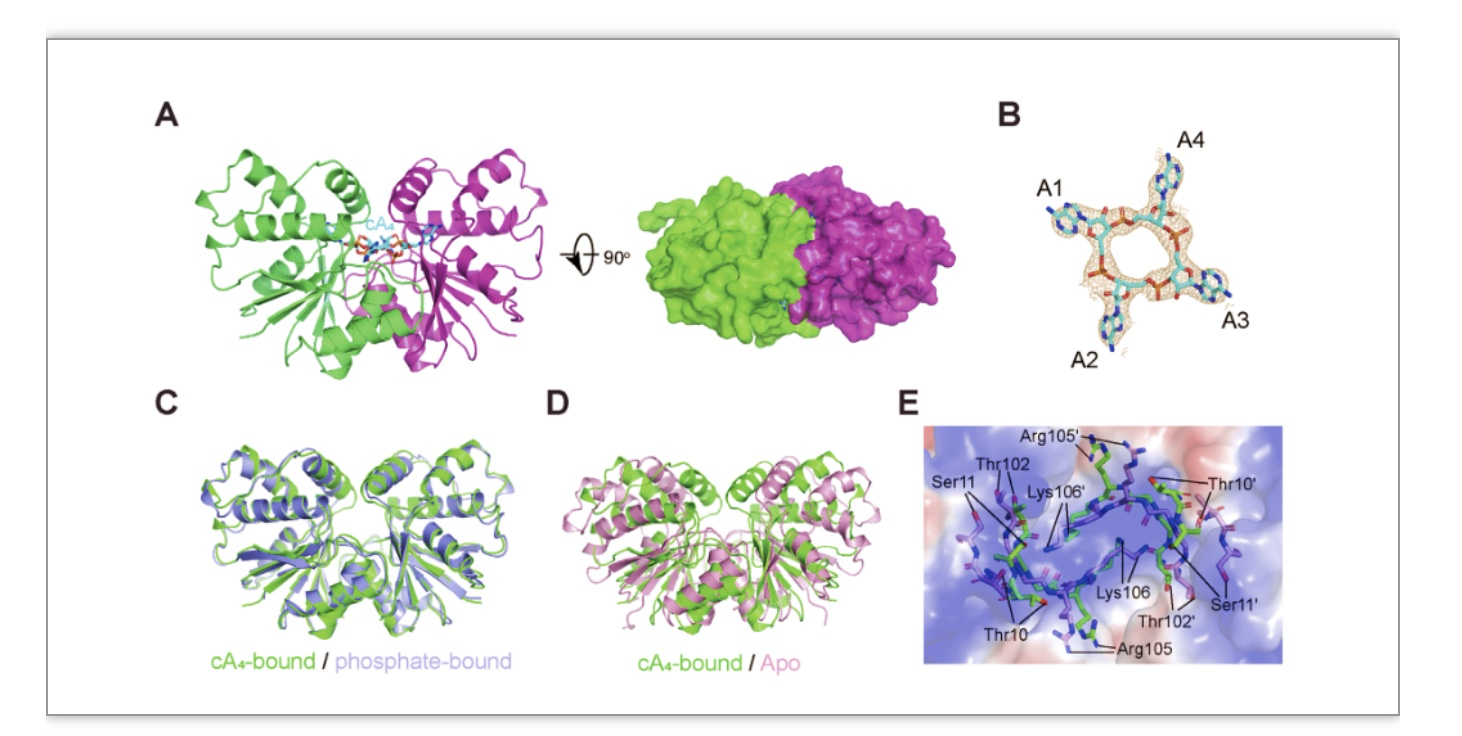
图2. Sso2081与cA4结合状态下的晶体结构
(图源:Du L , et al., Nucleic Acids Res. 2023)
此外,该研究还结合HPLC-MS、微量热泳动分析(MST)等多种生物化学实验技术,详细阐明了Sso2081识别和切割底物的分子机制:cA4的结合促使催化中心发生显著的构象变化,特别是两个催化残基Thr10和Ser11发生了约5.0埃的位移(图2E),同时位于活性口袋中央的Lys106也调整了其侧链方向而与位于两个剪切位点的磷酸根结合。此外,通过对Sso2081与切割中间体的结构解析,发现Lys106和Tyr133对于稳定中间过渡态具有重要的作用(图3B)。
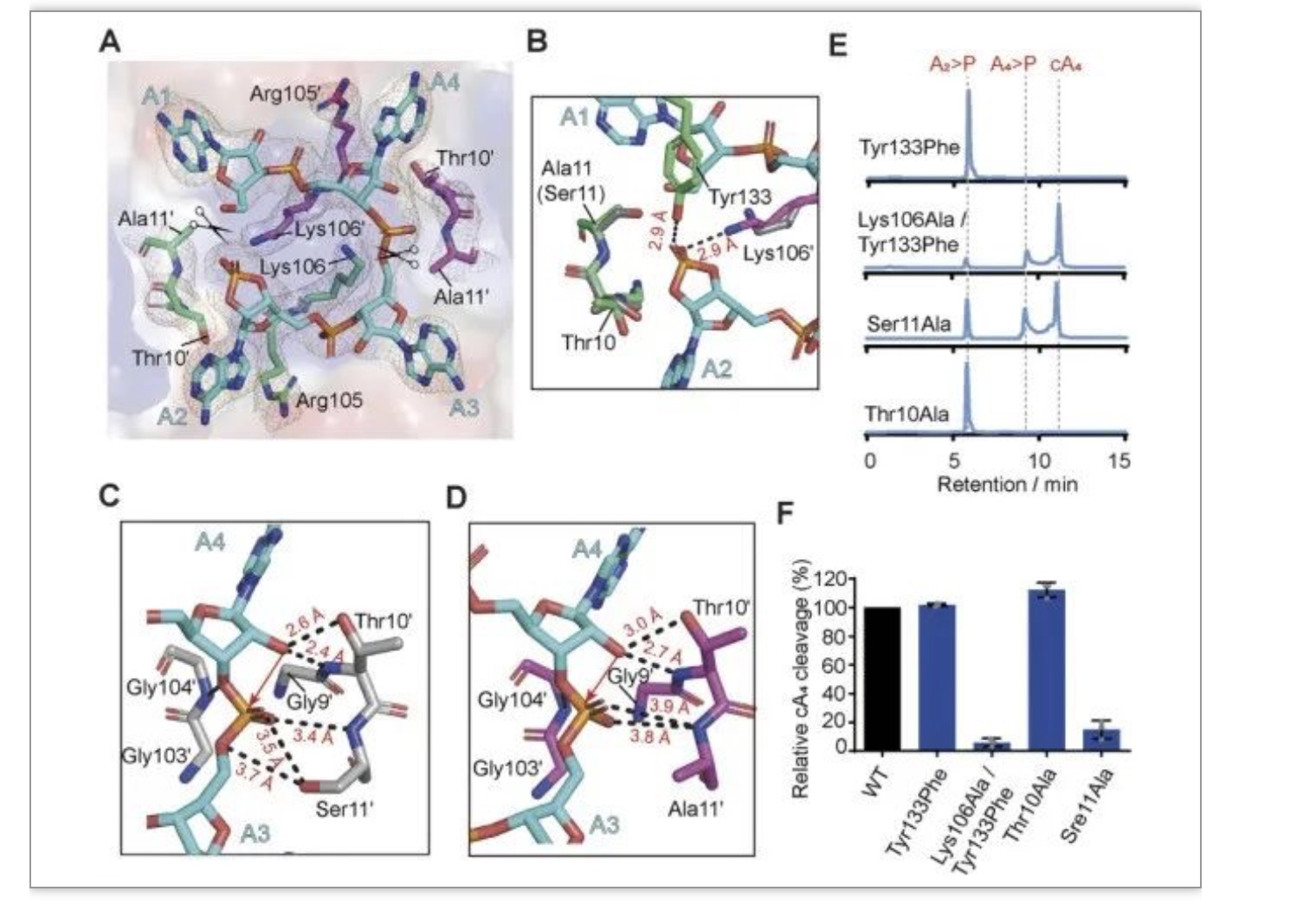
图3. Sso2081识别和切割cA4的分子机制
(图源:Du L , et al., Nucleic Acids Res. 2023)

