单细胞多组学测序技术使研究人员可以利用多种模态的数据来探索个体细胞的多样性和基因调控程序。单细胞RNA测序(scRNA-seq)和单细胞染色质转座酶可及性的高通量测序(scATAC-seq)可同时测量同一细胞中的转录组(transcriptome)和染色质可及性(chromatin accessibility)[1];转录组和表位的细胞标签测序(CITE-seq)可同时测得表面蛋白(surface protein)和转录组数据[2]。尽管整合单细胞多组学信息可以拓宽对基因组调控的理解,但高维、离散和稀疏的数据特性使该任务极具挑战性。现有的大多数单细胞数据分析方法要么局限于单一模态,要么无法处理模态缺失和缺乏可解释性。不仅如此,现有大多数方法是完全数据驱动的,没有充分利用基因注释或生物路径等先验知识。
本研究提出了一种基于编码主题模型的深度学习方法(Multi-Omics Embedded Topic Model),旨在处理高维度的单细胞多组学数据。moETM的主要目标包括实现细胞聚类、细胞子类型的识别、跨组填补以及细胞类型特征和生物标志物的鉴定。
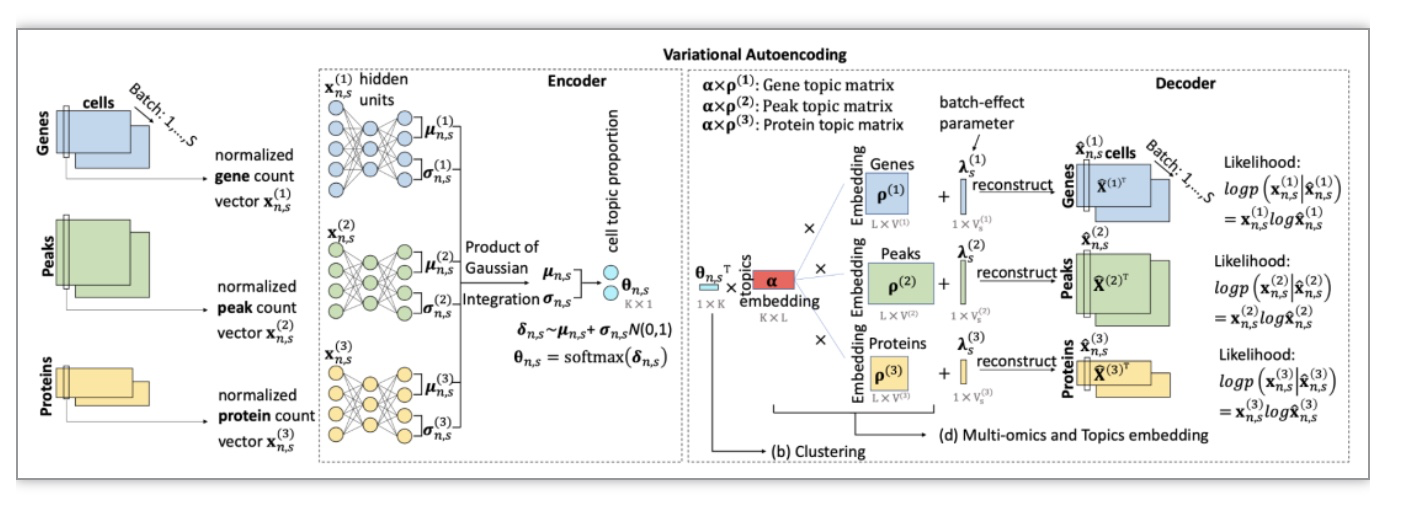
moETM建立在变分自动编码器(Variational Autoencoder)[3]之上。为了使VAE框架适用于单细胞多组学数据,该研究在VAE的编码器和解码器上主要做出了两项改进。在编码器中,研究者假设每个模态的潜在表征都遵循一个独立的K维逻辑高斯分布(logistic normal distribution),研究者采用了K维高斯的乘积,即高斯乘积(Product of Gaussians),以将这些分布有效地组合成多组学数据的联合分布。在解码器方面,moETM借鉴了早期的工作中线性矩阵分解的方法[4],从细胞特征编码(cell embedding)中重新构建表达矩阵。具体来说,解码器将细胞特征矩阵分解为与细胞样本无关的细胞主题矩阵、与细胞样本无关的主题编码矩阵和与细胞样本有关的特征编码矩阵。由于不同的模态共享相同的细胞主题矩阵,但有各自的细胞特征编码矩阵,研究者便可以以一种高度可解释的方式探索细胞、多组学主题和细胞特征之间的关系。
图1 moETM模型
为了展示moETM的实用性,研究者将其与现有最前沿的6种计算方法在7个数据集上进行了对比。在所有数据集中,moETM在包括保留生物变异(ARI和NMI)和去除批次影响(kBET[5]和GC)的4个常见评估指标上均表现出优异的性能。研究者还通过消融实验和可视化,进一步确认了在细胞聚类方面使用多模态的优势。除此之外,moETM经过训练后能完成跨组学的填补任务(cross-omic imputation)。比如moETM可准确地从基因表达数据中填补表面蛋白质表达,也可准确地从蛋白质表达数据中填补基因组学数据。前者为从高维组学到低维组学,后者为从低维模态到高维模态。虽然后者更有挑战性,moETM依旧取得了比现有方法更优异的表现。
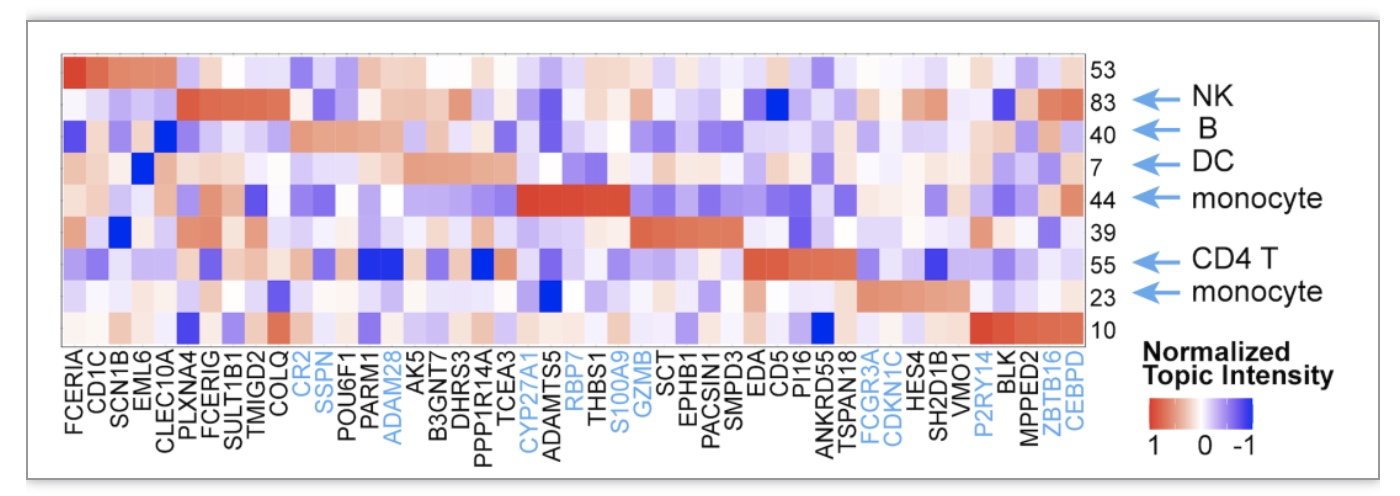
为探索moETM的解释性,作者还深入研究了moETM学习到的多组学主题和主题分数,并通过统计学分析或富集分析将其与细胞类型和生物路径连接。例如,在骨髓单核细胞(转录组+表面蛋白)数据集中,主题40里B细胞的主题分布显著高于其他细胞类型, CR2基因的主题分数高于其他基因。此外,CR2基因不仅是CellMarker数据库[5,6]中B细胞的标记基因,也被包含在该主题下富集的B细胞相关的基因集合里[5-7]。再者,以往文献显示[8],通过结合C3d,CR2可以降低在适应性免疫应答中B细胞激活的阈值,更进一步充分证实主题40下细胞类型和基因特征的统一性。
图2 骨髓单核细胞(转录组+表面蛋白)CITE-seq数据的主题分析
为了提高模型学得主题的可解释性,研究者进一步将先验知识,如基因集或生物路径,融入模型。研究者使用了大约7000个基因本体生物过程(GO-BP),在骨髓单核细胞(转录组+染色质可及性)数据集上训练了生物路径引导的p-moETM。从定量上来看,先验生物信息引导的p-moETM可在细胞聚类性能上达到与moETM相近的水平。
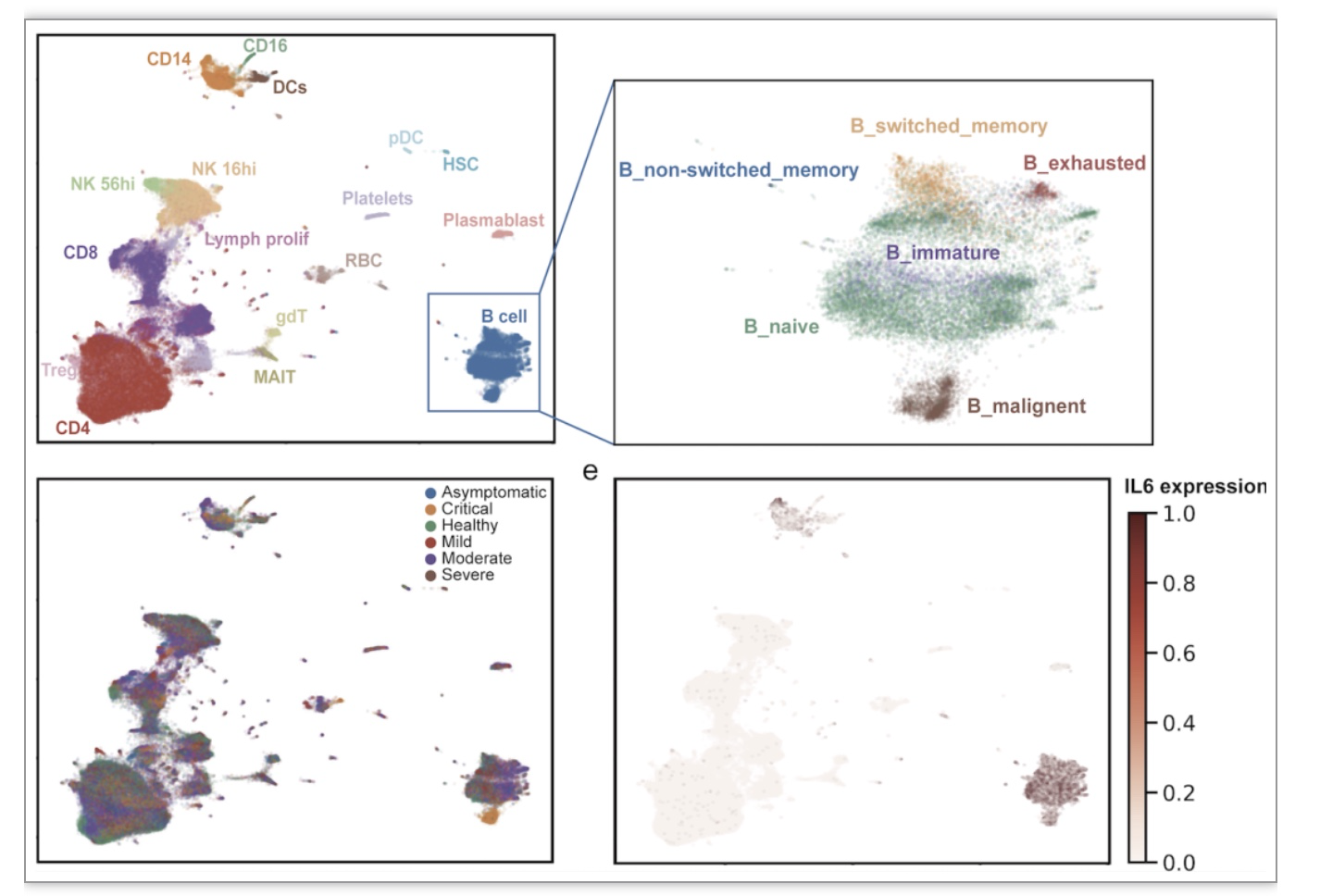
为将moETM拓展到临床解释上,该研究分析了COVID-19 CITE-seq数据集(转录组+表面蛋白)[9],并将moETM学到的免疫特异性主题和患者的严重状况联系起来。moETM的主题分析不仅揭示了免疫细胞相关的标记基因,还揭示了与COVID-19表型条件相关的细胞类型。该研究发现危重状态的患者展现出高主题概率的B恶性细胞。此外,B恶性细胞标记基因IL6在这些患者中与轻微症状和无症状患者相比表达有显著差异。
图3 COVID-19 CITE-seq数据的主题分析
综上所述,该文章开发了一个统一的可解释深度学习模型moETM,以用于整合包括转录组、染色质可及性和表面蛋白质在内的单细胞多组学数据。moETM可用于多模态整合、跨组学填补、以及识别细胞类型特定的主题、基因特征、表面蛋白质特征和调控网络基序。该研究还将moETM应用于COVID-19患者的CITE-seq数据分析,不仅揭示了已知的免疫细胞类型的基因特征,还提供了与COVID-19临床重要条件相关的生物标志物,从而从生物学和临床角度提供了见解。
在moETM 中存在一些未解决的挑战。例如,moETM 要求训练数据中所有组学数据都在同一细胞中测量。最近的一些研究利用图来学习和整合多组学单细胞数据,但这牺牲了计算复杂度和可解释性。此外,moETM 的另一个扩展是在模型训练之前,使用语言模型对序列信息进行预训练建模。事实上,随着计算成本的降低,已经有基于大语言模型的大型单细胞数据建模、转录组语言理解和基于序列的基因表达预测等应用涌现。