腺苷-肌苷(adenosine-to-inosine, A-to-I)RNA编辑是一种重要的转录后修饰,由腺苷脱氨酶(adenosine deaminase acting on RNA enzymes, ADARs)催化,通过改变RNA分子中的特定核苷酸使转录组多样化[1]。A-to-I编辑的失调在自身免疫性疾病、心血管疾病以及癌症中发挥重要作用[2]。已有研究全面、系统地解析了多种癌症相关的RNA编辑事件[3]。近年来,RNA编辑事件被证明可以影响癌症治疗的药物反应[4, 5],然而耐药相关的RNA编辑全景图谱及其影响耐药的分子机制的工作仍然匮乏。该研究系统地识别了肿瘤患者和细胞系中耐药相关的RNA编辑,揭示了RNA编辑在肿瘤耐药中的全景图谱及其调控机制,为肿瘤耐药性研究提供了新的治疗靶点和重要参考。

研究人员基于六种癌症患者的RNA编辑数据以及患者对18种药物的药物反应数据,从98127个RNA编辑位点中识别出了7157个差异编辑位点(differential editing sites, DESs)。同时,研究人员还基于CCLE(Cancer Cell Line Encyclopedia)和GDSC(Genomics of Drug Sensitivity in Cancer)的细胞系数据识别出了DESs。结果表明,在相同的状态(一种药物和一种癌症的组合被定义为一种状态)下,患者和细胞系的DESs存在显著富集。此外,研究人员发现大部分DESs与患者的预后显著相关,且DESs在耐药患者和预后差的患者中呈现出一致的编辑模式(图1),例如,位于METTL7A的3’非翻译区(3’-untranslated region,3’-UTR)的位点chr12:51324627,在耐药患者和预后差的患者中均呈现显著的高编辑。
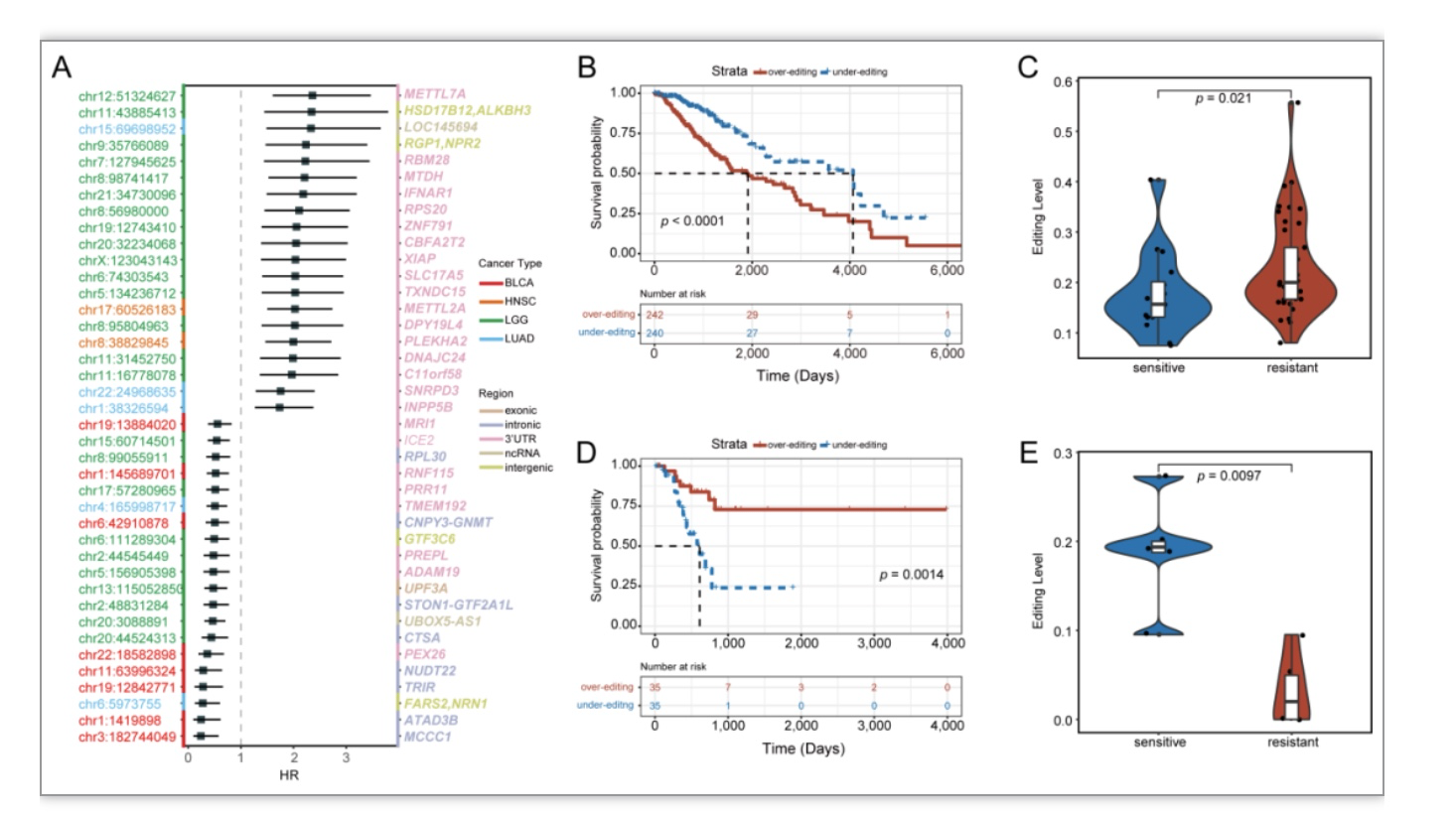
图1 DESs与预后相关
(图源:Xu Zhou et al., Advanced Science, 2023)
为了阐明DESs参与肿瘤耐药的作用机制,研究人员分析了DESs的编辑特征,发现DESs在不同状态下的耐药样本中呈现出多种编辑模式,进一步发现ADARs的表达水平并不能完全解释这些编辑模式(图2)。因此,研究人员基于DESs附近的motif信息预测了调控DESs的RNA结合蛋白,其中一些RNA结合蛋白已经被证实能够影响RNA编辑,例如TARDBP[6],还有一些RNA结合蛋白也已经被证实与肿瘤耐药相关,例如FXR1[7]。这些结果表明耐药相关的RNA结合蛋白可能通过与ADARs互作来调节RNA编辑事件。
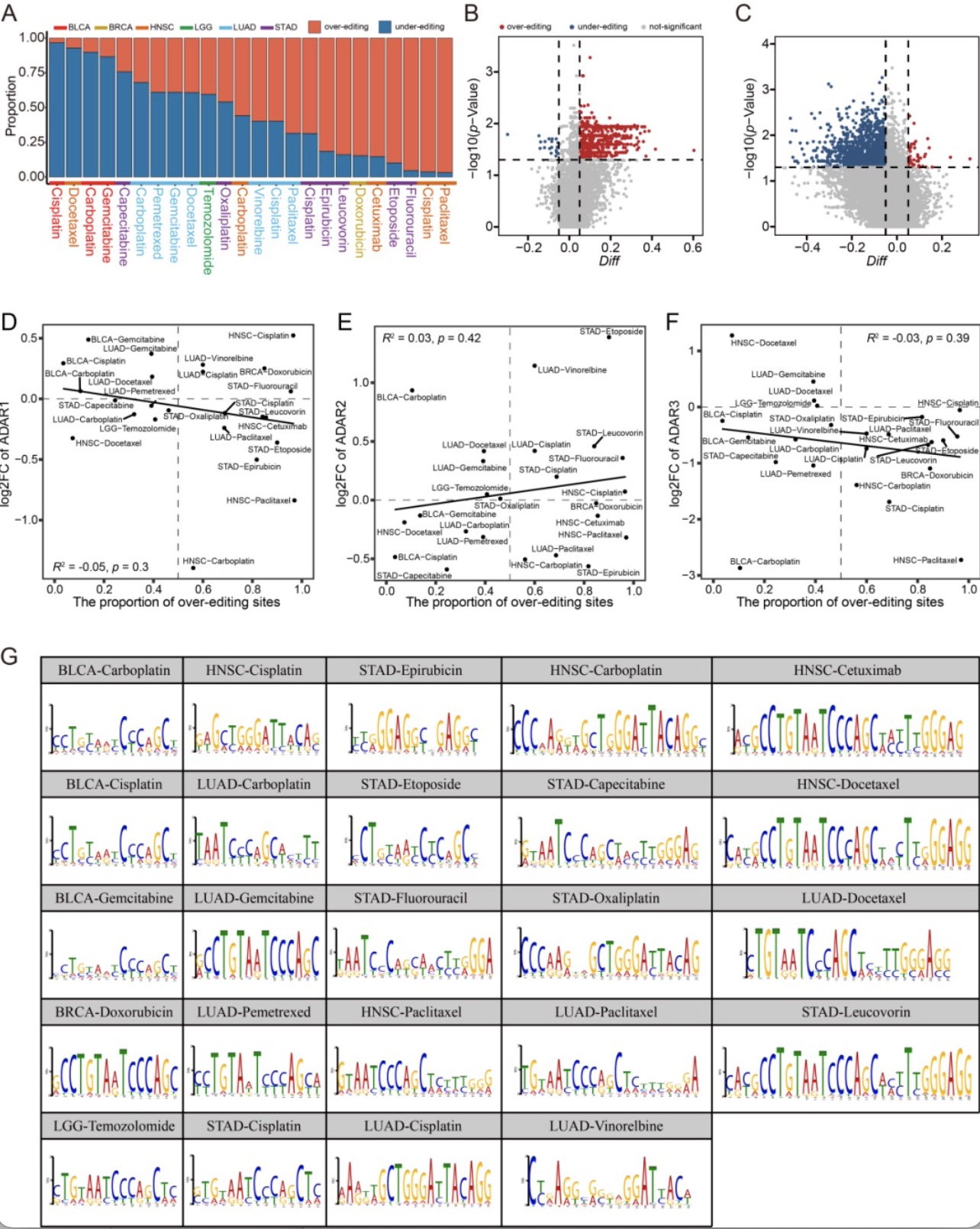
图2 RNA编辑模式及RNA结合蛋白的预测
(图源:Xu Zhou et al., Advanced Science, 2023)
在肿瘤患者和细胞系中,研究人员都发现DESs显著富集在3’-UTR区域。因此,研究人员进一步揭示了3’-UTR上的DESs通过介导miRNA调控从而影响肿瘤细胞的耐药性,并识别出一些潜在的介导肿瘤细胞耐药的DES-miRNA-基因三元组(图3)。
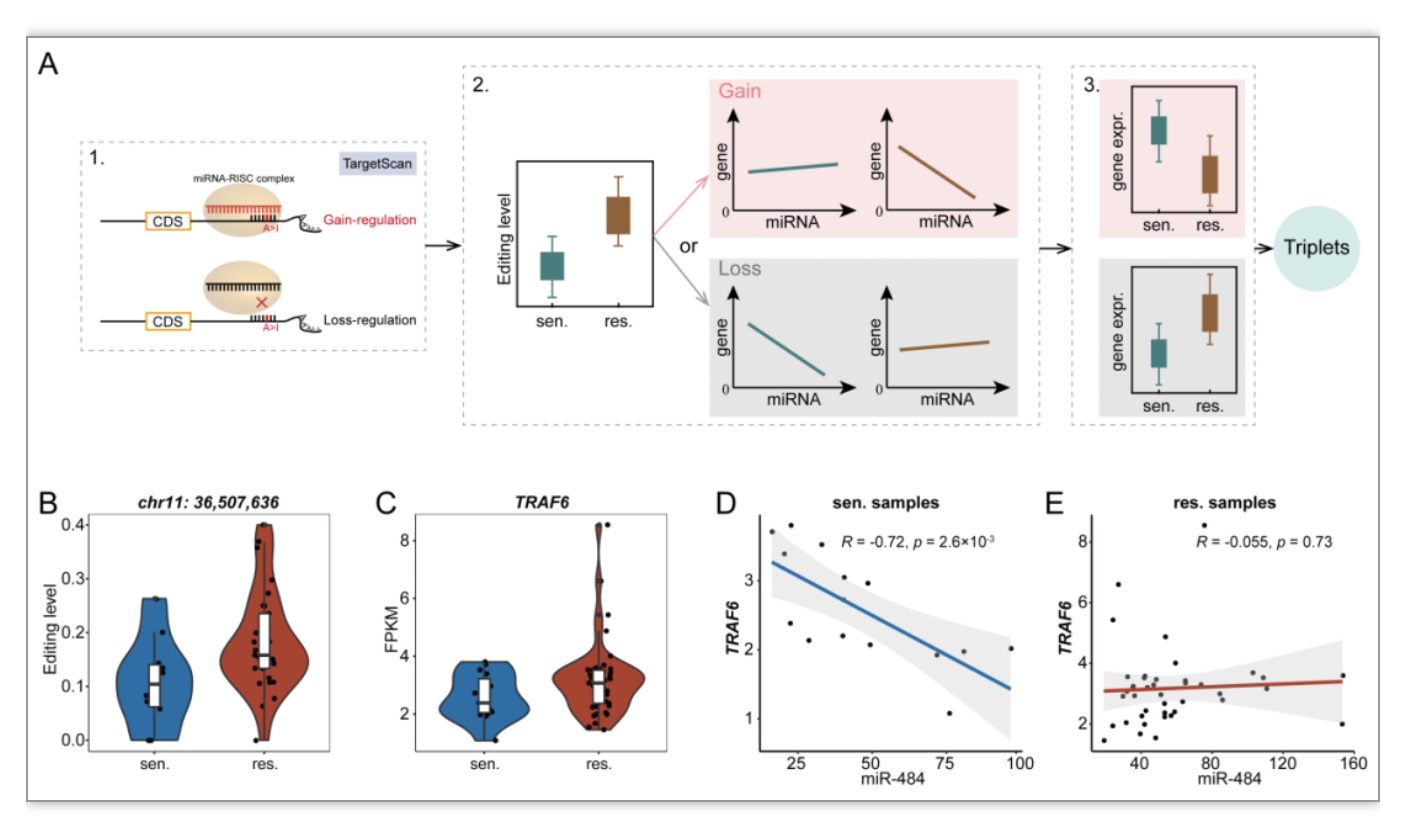
图3 DESs影响miRNA调控
(图源:Xu Zhou et al., Advanced Science, 2023)
最后,该团队搭建了在线数据库REDR(http://www.jianglab.cn/REDR/),支持用户检索耐药相关的RNA编辑及其影响的miRNA调控信息(图4)。用户可以在Search界面输入癌症类型、药物和基因查询相应状态下患者和细胞系中的DESs信息,或通过Genome Browser直观浏览和查询DESs信息;用户还可以输入基因或miRNA查询DESs影响的miRNA调控信息(三元组),或在Triplet界面浏览DESs影响的miRNA调控信息。
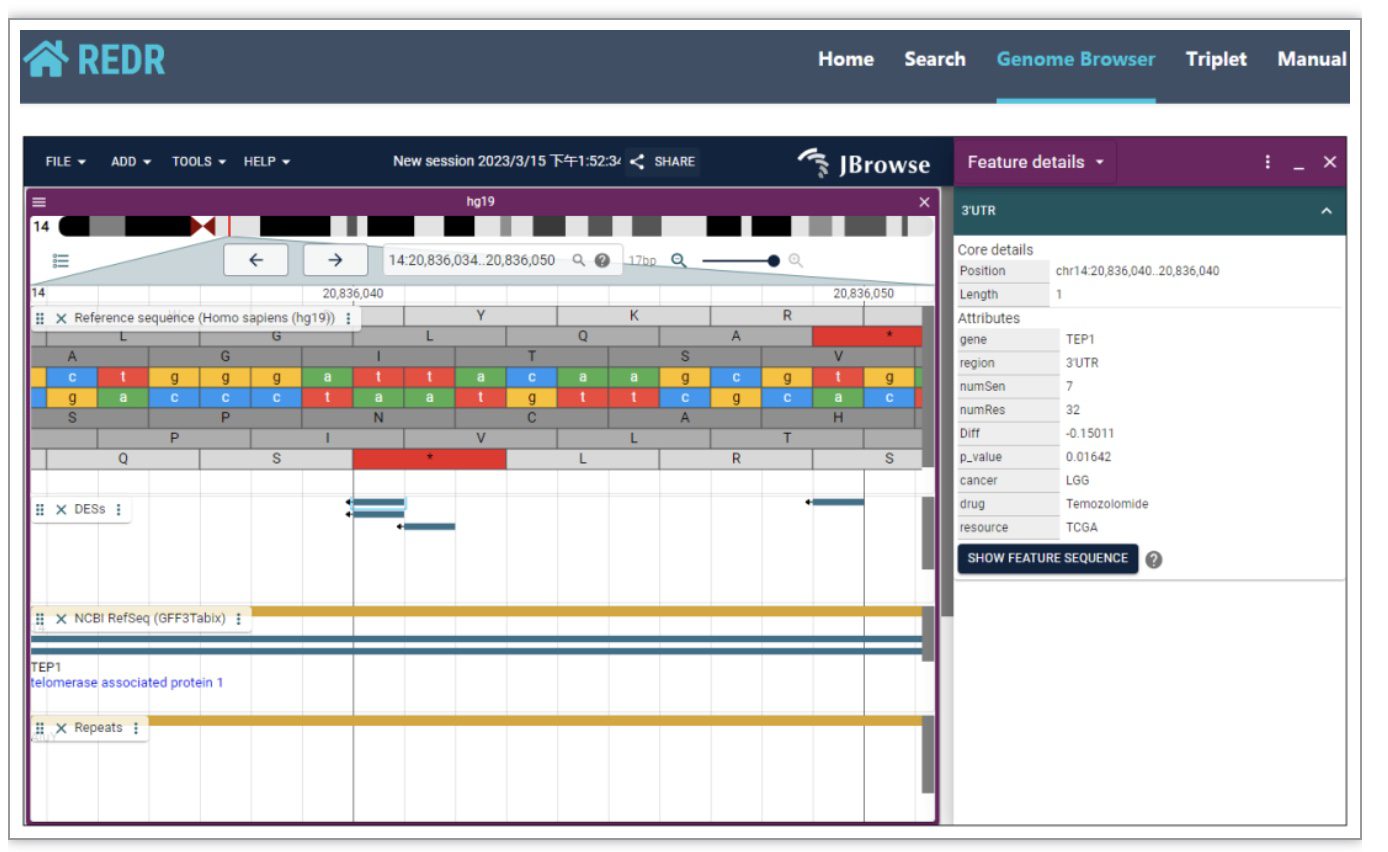
图4 REDR数据库
(图源:Xu Zhou et al., Advanced Science, 2023)

