前列腺癌 (prostate cancer, PCa) 是男性中第二大常见肿瘤,在全球范围内影响着数百万人。据估计,仅2020年全球有超过141万男性被诊断出患有前列腺癌,并且有37万多男性死于前列腺癌[1]。前列腺癌已成为严重威胁全球中老年男性健康的疾病,而且其发病率有年轻化趋势。在我国, 前列腺癌的发病率及死亡率均呈快速加剧趋势。前列腺癌的发病机制复杂,年龄、种族、及家族遗传等都会增加患病风险。而在所有风险因子中,遗传度可达57%[2]。全基因组关联分析(GWAS)是指通过对疾病组与正常组间的比较在人类全基因组范围内找出特定性状或疾病相关的序列变异,即单核苷酸多态性(SNP)位点。随着GWAS研究的不断拓展和深入,前列腺癌遗传易感性得到了很好的揭示。因此,深入认识并阐明风险SNP对前列腺癌的发病机制研究和治疗方法的探究十分重要。
“Extensive germline-somatic interplay contributes to prostate cancer progression through HNF1B co-option of TMPRSS2-ERG” 的研究。该研究发现转录因子基因多富集于前列腺癌遗传风险基因座,其中17q12/HNF1B位点的多个SNP通过TMPRSS2-ERG (T2E) 融合转录因子调控HNF1B的表达,从而加速前列腺癌细胞的生长并促进肿瘤进展。此外HNF1B协同T2E融合基因介导多个风险相关基因座,包括17p13.3/VPS53/FAM57A/GEMIN4的生物学效应。该研究揭示了胚系遗传变异与体系T2E融合基因间广泛的相互作用影响前列腺癌风险和进展,为临床研究和治疗提供了新的标志物和潜在靶点,并为研究17q12/HNF1B等位点在其它肿瘤中的作用提供了范式。

已有研究表明转录因子通过与基因组顺式调控元件(CRE) 结合,在基因表达网络中发挥核心作用[3,4]。由此,作者发现包括HNF1B在内的转录因子基因在前列腺癌易感基因座中显著富集(图1a-b)。这些前列腺癌风险位点中,HOXB13、RFX6和NKX3-1已被证明与前列腺癌易感性和肿瘤发生存在因果关系[5-8]。而在功能上尚未表征的风险基因座中,17q12/HNF1B区域的多个SNP被多次报道与前列腺癌易感性相关[9-16]。作者通过表型组大数据关联分析(PheWAS)再次证实17q12/HNF1B变异与前列腺癌显著关联(图1c),结合其他研究关于17q12/HNF1B与多种癌症类型相关的报道[17-21],促使作者深入研究该基因座的分子和生物学机制及其临床意义,以及HNF1B在前列腺癌中的作用。
作者首先进行了细胞增殖实验,并发现敲低HNF1B减弱了肿瘤细胞的增殖和迁移(图1d-f)。此外,全基因组CRISPR介导的功能缺失筛选的数据表明HNF1B是前列腺癌细胞存活的必需基因(图1g),进一步表明HNF1B在前列腺癌细胞生长和存活的重要性。为了研究HNF1B与前列腺癌临床相关性,作者分析了大量肿瘤相关数据。结果显示,在原发性,转移性及在恶性程度更高的前列腺癌肿瘤中HNF1B的表达水平显著升高(图1h-k)。这些发现表明HNF1B在前列腺癌进展中具有潜在作用,是17q12 位点的潜在致病基因。
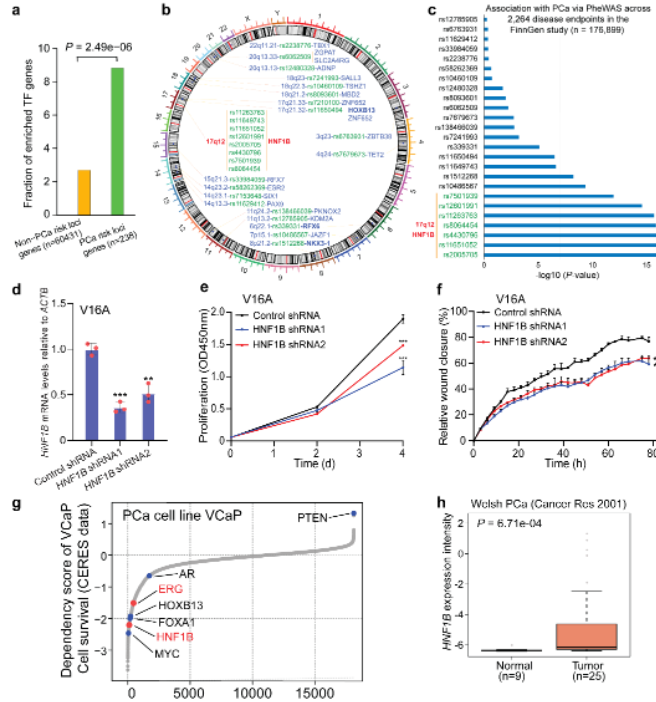
图1 转录因子基因在前列腺癌风险位点附近高度富集,17q12/HNF1B与前列腺癌细胞生长和肿瘤进展相关
(图源:Giannareas N, et al., Nat Commun, 2022)
紧接着,为了识别位于17q12基因座的功能性因果变异和确定风险相关基因,作者进行了表达数量性状位点(eQTL)分析,筛选了13个与前列腺癌风险高度相关的SNP,并发现其中11个SNP与HNF1B的表达存在显著关联(图2a)。并且在一兆碱基窗口内所有基因中,HNF1B是唯一的强关联基因(图 2b)。CRISPR/Cas9介导的基因组编辑删除了含SNP区域,结果表明HNF1B的表达在大多数混合克隆中显着下调(图2c)。随后的增强子报告基因实验表明,多个含SNP区域显示类增强子功能(图2d)。另外CRISPR/Cas9介导的SNP增强子区删除的单细胞克隆结果同样验证了HNF1B表达水平显著降低(图2e)。这些结果表明HNF1B是前列腺癌风险相关基因,并发现了该基因座多个潜在致病SNP。
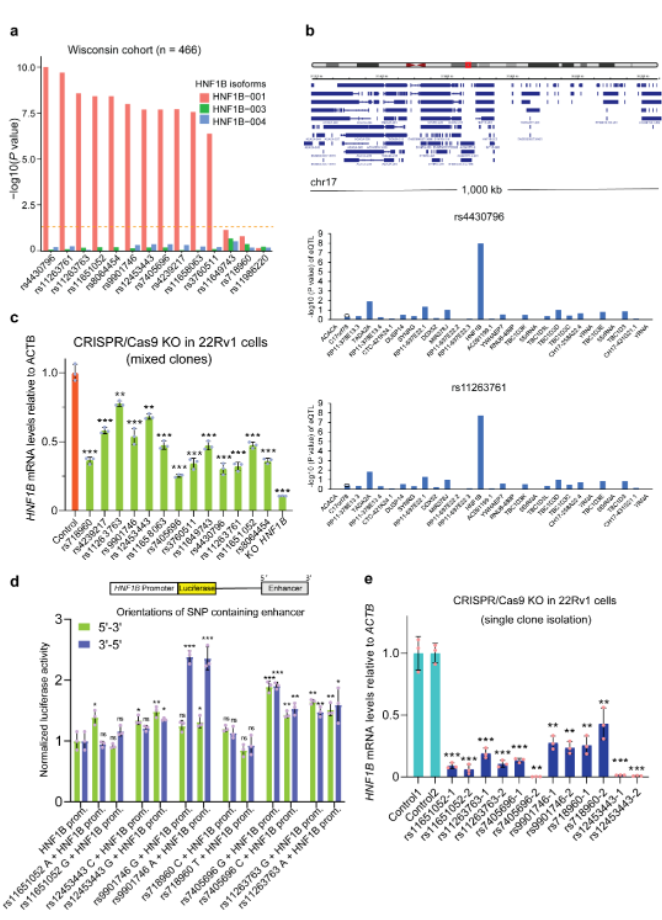
图2 17q12位点多个 eQTL SNP是潜在的因果变异并参与调节 HNF1B的表达
(图源:Giannareas N, et al., Nat Commun, 2022)
作者推测可能有潜在的转录因子调控17q12/HNF1B位点,多个大型临床前列腺癌样本均显示ERG为HNF1B最强相关共表达基因(图3a-b),而且敲低ERG可导致HNF1B表达降低(图3c)。为探索HNF1B基因座的调控机制,作者分析了大量全基因组染色质免疫沉淀测序(ChIP-seq)数据,发现表观遗传标记(H3K4me1/2和H3K27ac)及ERG大量富集于HNF1B基因座附近的多个潜在因果SNP区域,并得到ChIP-qPCR验证(图3d-i)。作者在人前列腺肿瘤样品中发现ERG和HNF1B之间的表达相关性取决于T2E融合状态。eQTL分析结果显示,17q12风险SNP仅在T2E融合阳性肿瘤组中与HNF1B表达显著相关(图3l-n)。这些结果表明,该位点遗传变异对靶基因HNF1B的影响是通过T2E基因融合来协调的。
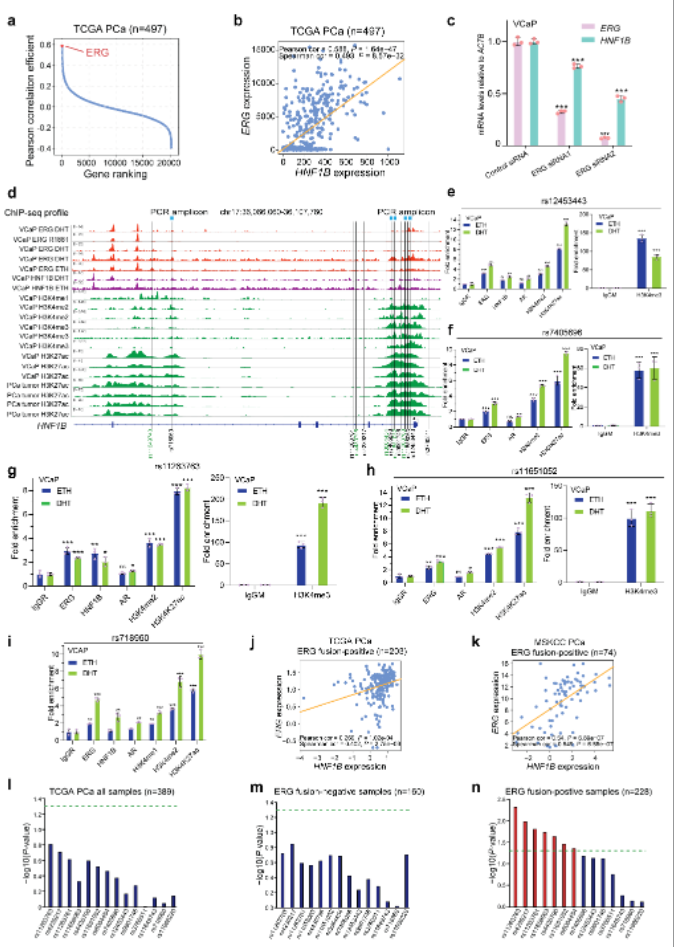
图3 ERG是17q12基因座的转录调节因子,并以依赖于TMPRSS2-ERG融合状态的方式在前列腺癌细胞和组织中调控HNF1B
(图源:Giannareas N, et al., Nat Commun, 2022)
为研究HNF1B在前列腺癌中的作用,作者在VCaP细胞中敲低HNF1B并进行了RNA-seq分析,结果表明HNF1B上调差异基因富集在与细胞周期相关的通路中,进一步支持了HNF1B促进细胞增殖和侵袭的发现。为了探索 HNF1B靶基因在前列腺癌中的临床相关性,作者生成了HNF1B靶基因特征集,并发现其在临床上与肿瘤进展和严重程度相关(图 4a-r)。

图4 HNF1B基因特征集在临床上与前列腺癌进展高度相关
(图源:Giannareas N, et al., Nat Commun, 2022)
作者进一步做了免疫共沉淀(Co-IP)实验,发现HNF1B和T2E之间存在蛋白相互作用(图5a-d)。同时ChIP-seq实验发现,超过70%的HNF1B结合位点与T2E基因组结合区域重叠(图5e)。作者接下来整合了RNA-seq与ChIP-seq数据,分析得到51个HNF1B/ERG直接调控的靶基因(图5f-g), 并发现该基因集具有很强的临床相关性(图5h-k) 。这些结果表明HNF1B和T2E之间存在多层次胚系-体细胞突变相互协同作用,并影响前列腺癌进展和预后。
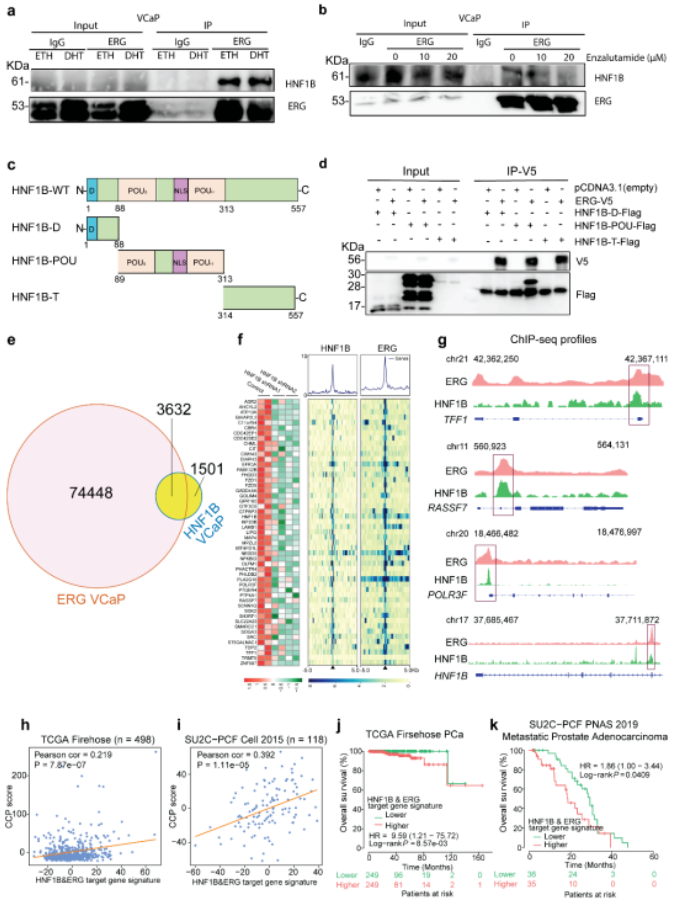
图5 HNF1B与ERG蛋白相互作用,并与ERG染色质共结合调节前列腺癌临床相关基因表达
(图源:Giannareas N, et al., Nat Commun, 2022)
鉴于以上发现的HNF1B与ERG多层次关联作用,作者设想HNF1B或与ERG一起影响前列腺癌遗传风险。相关分析发现,前列腺癌风险SNP更倾向于富集在ERG和HNF1B的共同基因组结合位点(图6a),共包括5个前列腺癌风险位点,且证实其关联的eQTL基因在临床上具有预后价值(图6b-d)。基因共表达分析表明17p13.3位点的三个eQTL基因FAM57A,GEMIN4和VPS53与HNF1B和ERG 高度共表达(图6e)。由此作者专注于该17p13.3/rs684232基因座,且已被重复报道与前列腺癌易感性密切相关,但其潜在的生物学和功能机制仍然未知 [22,23] 。ChIP-seq结果显示HNF1B和ERG在该位点有多个强结合峰,且rs684232可影响HNF1B和ERG的DNA结合基序(图6f-g),进而表明其可潜在影响HNF1B和ERG的转录调节。ERG和HNF1B敲低或敲除功能实验显示这三个基因的表达水平也随之下调(图6h-i) 。人前列腺肿瘤数据显示FAM57A与ERG或HNF1B表达呈高度正相关,且FAM57A与HNF1B的表达水平依赖于前列腺肿瘤中的ERG融合状态(图6j-m)。这些结果表明T2E融合及HNF1B共同调节17p13.3风险基因。
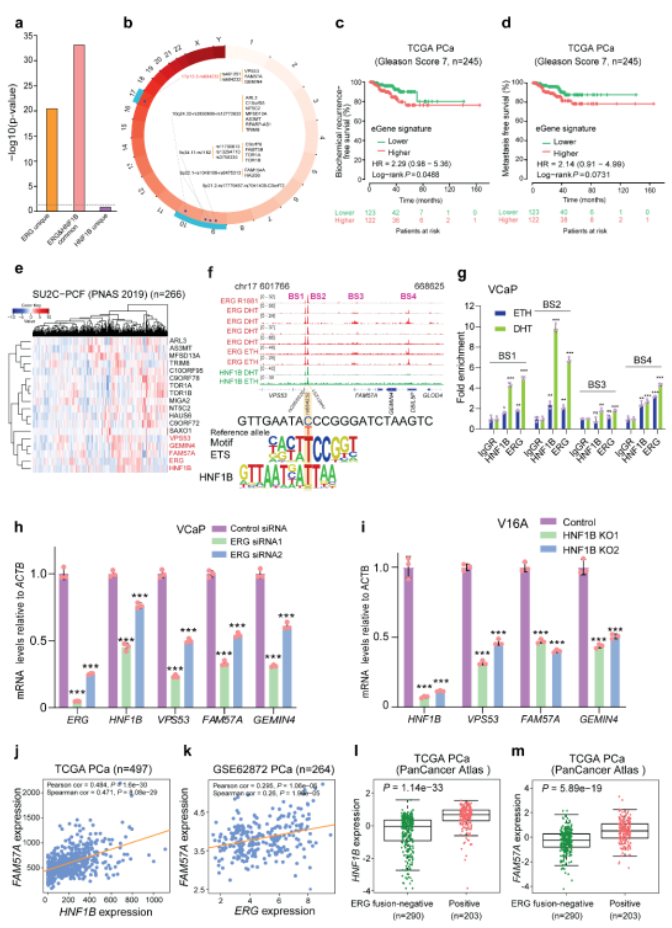
图6 前列腺癌易感性等位基因高度富集在HNF1B和ERG共同结合的染色质基因组区域
(图源:Giannareas N, et al., Nat Commun, 2022)
为进一步探究17p13.3区域是否具有增强子功能,作者进一步做了增强子报告基因分析,定量染色体构象捕获测定(3C-qPCR)和CRISPR/Cas9基因编辑等系列实验。结果表明17p13.3/rs2955626、rs684232和 rs461251均参与调节VPS53、FAM57A和GEMIN4的表达(图7a-c) 。为探究17p13.3基因座基因的生物学相关性,作者进行了肿瘤细胞相关实验。结果显示在敲低VPS53、FAM57A或GEMIN4后肿瘤细胞均显示出显著减弱的细胞增殖与迁移力(图7d-j)。这些数据表明,17p13.3是个多因果变异和多致病基因位点,协同增强前列腺癌易感性和肿瘤细胞转化。
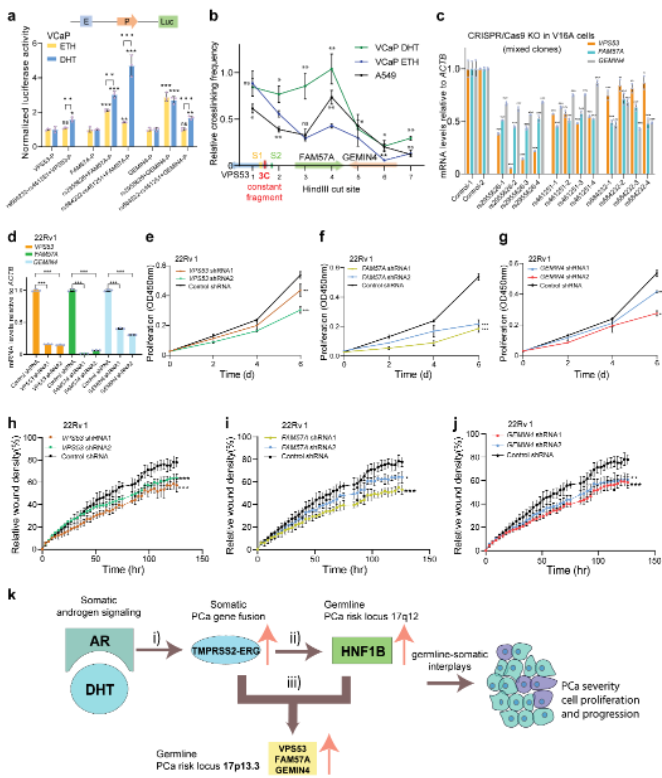
图7 前列腺癌17p13.3 易感性位点功能SNP参与调控VPS53、FAM57A和GEMIN4表达和功能
(图源:Giannareas N, et al., Nat Commun, 2022)

