摘 要:RNA 可以被100 余种化学修饰所修饰。这些化学修饰以甲基化为主,广泛分布于各种类型的RNA中,如rRNA、tRNA、snRNA、snoRNA 和mRNA 等,其中针对mRNA 内部修饰丰度最高的6- 甲基腺嘌呤(m6A) 的研究最为深入。m6A 修饰酶( 甲基转移酶METTL3/METTL14/WTAP 和去甲基化酶ALKBH5 与FTO) 和结合蛋白YTHDF2、YTHDF1 与YTHDC1 的发现,证明了RNA 甲基化修饰同DNA甲基化修饰一样是动态可逆的,从而将RNA 甲基化修饰由微调控机制提升到表观转录组新层次。而候选m5C 修饰甲基转移酶NSUN 家族蛋白和去甲基化酶TET 蛋白的初步鉴定,丰富了RNA 甲基化修饰表观转录组研究内涵。RNA 甲基化介导的表观转录组学调控和作用已成为RNA 生物学新研究领域。现重点回顾和展望RNA 的m6A 和m5C 甲基化修饰特征及其潜在生物学功能。
关键词:6- 甲基腺嘌呤(m6A) ;5- 甲基胞嘧啶(m5C) ;mRNA ;非编码RNA ;甲基转移酶;去甲基化酶;甲基化结合蛋白
中图分类号: Q523 ;Q752 文献标志码:A
Dynamics and biological significance of RNA methylations
SUN Hui-Ying1,2#, HAO Ya-Juan1,2#, PING Xiao-Li1,2, HUANG Chun-Min1, YANG Ying1, SUN Bao-Fa1, YANG Yun-Gui1*
(1 Key Laboratory of Genomics and Precision Medicine, Collaborative Innovation Center of Genetics and Development, Beijing Institute of Genomics, Chinese Academy of Sciences, Beijing 100101, China; 2 University of Chinese Academy of Sciences, Beijing 100049, China)
Abstract: Over 100 types of chemical modifications have been identified in various types of RNAs including rRNA, tRNA, snRNA, snoRNA and mRNA, among which methylation is most common. The N6-methyl-adenosine (m6A) as the most common and abundant internal modification on mRNA molecules has been widely studied. The recent identification of m6A modifying enzymes including methyltransferase complex METTL3/METTL14/WTAP, two demethylases ALKBH5 and FTO and binding proteins YTHDF2, YTHDF1 and YTHDC1, indicates that RNA methylation is reversible and represents a novel epitranscriptomic mechanism instead of micro-regulation in gene expression control. Identification of candidate m5C modifying enzymes NSUN family proteins and TET proteins further expands RNA methylation-mediated epitranscriptomics. Epitranscriptomics has become a new research frontier in RNA biology. This review summarizes recent progress in RNA m6A and m5C methylations and proposes their potential biological significance.
Key words: m6A; m5C; mRNA; non-coding RNA; methyltransferase; demethylase; methylation binding protein
RNA 存在100 余种化学修饰[1],甲基化是主要的修饰形式,6- 甲基腺嘌呤(m6A) 和5- 甲基胞嘧啶(m5C) 是其中最具有代表性的两种修饰。m6A甲基化修饰于20 世纪70 年代被发现,随后的研究陆续证实了该修饰广泛存在于包括动植物、原核生物以及病毒在内的多个物种中[2-8]。近年来,随着酶学技术的发展,m6A的修饰酶相继被发现,其中WTAP、METTL3和METTL14复合物可以催化m6A的形成,而FTO和ALKBH5可以使其去甲基化;m6A 修饰的生物学功能可能主要通过包含有YTH结构域的结合蛋白来发挥[9]。m6A 修饰如何调控编码基因mRNA 和非编码RNA 加工代谢目前尚不明确。m5C 修饰也存在于mRNA、tRNA、rRNA 及长非编码RNA 中。NSUN 家族蛋白,如NSUN2、NSUN4和DNMT2 是候选m5C 甲基转移酶[10-12]。下面以m6A 和m5C 修饰为主线,从各自的主要检测技术、分布特征、甲基化/ 去甲基化酶及结合蛋白、生物学功能等进行阐述。
1 6-甲基腺嘌呤:m6A
1.1 检测技术
早期研究主要通过化学手段,包括液相色谱技术检测m6A 含量,它占mRNA 中腺嘌呤总量的0.1%~0.4%[13-14],并发现其修饰区域倾向于包含“RRACH”([G/A][G>A]m6AC[U>A>C]) 的保守序列[7]。近年,两个独立研究团队结合基于m6A 抗体免疫共沉淀和高通量测序技术(meRIP-seq 和m6Aseq技术),获得了转录组水平上的m6A 修饰数量和分布特征[15-16]。多个实验室对m6A 单碱基分辨率技术开发进行了有益的尝试,包括基于修饰碱基回补温度差异法[17]、基于RNA 酶切割位点特异性并结合放射性元素标记及薄层层析方法的SCARLET (site-specific cleavage and radioactive-labeling followed by ligation-assisted extraction and TLC)[18] 和光交联-m6A 抗体免疫共沉淀联用法,但m6A 修饰位点的定性和定量化还需要进一步优化。
1.2 分布和保守性
m6A 倾向于分布在mRNA 的蛋白质编码序列(CDS)、3′ 非翻译区(3′UTR)、终止密码子附近、剪切位点附近以及长的外显子区域,而且其修饰区域在人和鼠中具有很高的保守性;在microRNA 靶位点及其邻近区域也有m6A 富集[15-16,19]。
1.3 m6A修饰酶及结合蛋白(图1A)
m6A 甲基转移酶复合物METTL3、METTL14和WTAP能以S-腺苷甲硫氨酸(S-adenosyl-L-methionine, SAM) 为甲基化供体,催化RNA 的m6A 形成,其中WTAP 作为调节亚基调控催化亚基METTL3 和METTL14 细胞内活性[20-25]。FTO 和ALKBH5 在二价铁离子和2- 酮戊二酸的辅助下催化m6A 去甲基化[26-27]。含有YTH 功能结构域家族的蛋白YTHDF2和YTHDC1 能结合m6A 甲基化修饰序列[8-9,15]。
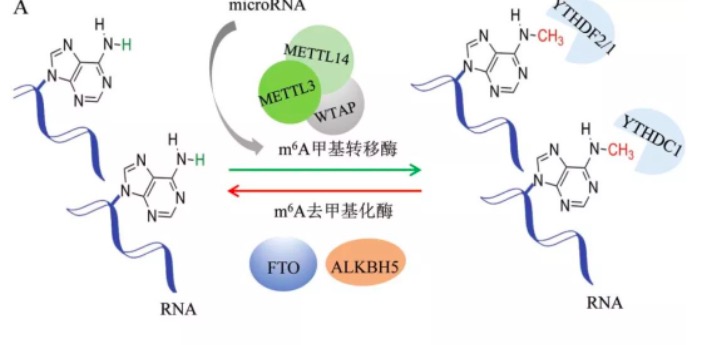
A:mRNAm6A修饰的动态可逆过程。m6A甲基转移酶复合体亚基METTL3/METTL14/WTAP 催化A形成甲基化的m6A;去甲基化酶FTO、ALKBH5催化其去甲基化;YTH家族蛋白YTHDC1结合细胞核内的m6A修饰,YTHDF2/1结合细胞质的m6A;microRNA通过调节METTL3的活性及结合RNA的能力调控m6A生成。
B:m6A修饰调控mRNA加工代谢潜在功能。前体premRNA转录后需要经过剪接、出核运输、定位、翻译和降解(稳定性)等加工和代谢,而m6A作为mRNA内部的重要可逆化学修饰,可能作为新顺式元件对上述进程进行调控。
图1 RNA m6A修饰的动态可逆过程及调控mRNA加工代谢的潜在功能
1.3.1 m6A甲基转移酶(编码器)
催化m6A 甲基化形成的甲基转移酶于1992年通过分离纯化首次被发现[28],前期研究将HeLa 细胞核提取物与人工合成的RNA 寡核苷酸片段共同孵育分离得到了两种组分,分别命名为MT-A 和MT-B,它们协同催化m6A 形成[29]。随后的研究分离得到了3 个组分MT-A1、MT-A2 和MT-B[29]。在设定的反应条件下,相对分子质量为200 kDa 的MT-A2 和800 kDa 的MT-B 两个组分具备了较强催化m6A 形成酶活性,而只有相对分子质量为30 kDa 的MT-A1 在m6A 甲基化形成的过程中作用相对较小[30]。
MT-A2 中70 kDa 的METTL3 亚基( 也称MTA70亚基) 蛋白预测的氨基酸序列中包含原核生物甲基转移酶的两个甲基化基序,分别是S- 腺苷甲硫氨酸(SAM) 结合位点和有催化功能的DPPW(Asp-Pro-Pro-Trp) 功能结构域[30]。在HeLa 细胞中,METTL3 表达缺陷会导致m6A 水平降低约30%。免疫荧光实验显示,METTL3 定位在富含剪切因子的细胞核内亚细胞器——核小斑(nuclear speckle)上,揭示了m6A 修饰可能和RNA 的剪切加工相关[24,30]。METTL3 在人体组织中广泛表达,并保守存在于多个物种中,如拟南芥中的MTA、果蝇中的IME4 等,在这些物种中的一些研究表明METTL3缺陷会引起减数分裂或发育异常[31-35]。
通过对METTL3 同源蛋白的分析,研究者发现METTL14 同样含有催化m6A 形成的S-腺苷甲硫氨酸结合位点和有催化功能的EPPL功能结构域,因此,METTL14 被认为是m6A 甲基转移酶复合体的另一亚基[36]。在催化m6A形成的过程中,METTL3和METTL14 按1:1 的比例形成二聚体直接相互作用,从而增强两者的甲基化催化能力[20,23]。光活性增强的核糖核苷交联和免疫共沉淀(photoactivatableribonucleoside-enhanced crosslinking and immunoprecipitation, PAR-CLIP) 结合二代高通量测序技术的数据分析表明,METTL14 和METTL3 含有相同的RNA 底物结合序列,且该序列包含已报道的m6A 经典保守基序RRACH[23]。同时,METTL14 与METTL3 一致定位在富含剪切因子的细胞核内亚细胞器——核小斑上,其定位暗示了m6A 的修饰可能会与RNA 加工过程中的可变剪切相关。
WTAP 与METTL3、METTL14 形成复合体,共同调控RNA m6A 的甲基化过程[24]。WTAP 首先结合到目标RNA 上,进而招募催化亚基METTL3和METTL14 形成的二聚体行使催化功能[20-22]。WTAP 最初作为剪切因子以及肿瘤结合蛋白被发现,后来被证实参与m6A 甲基化修饰、细胞周期以及哺乳动物胚胎细胞的发育等过程[37-38]。WTAP缺失不仅会导致m6A 水平下降,而且会引起结合到METTL3 上的RNA 减少[24],这一结果表明WTAP的作用可能是招募甲基转移酶的催化亚基METTL3和METTL14结合到RNA上,WTAP 同METTL3、METTL14 一样,定位在核内亚细胞器——核小斑
上,可以与两者形成复合体,加速m6A 甲基化的形成,进而调控m6A 的动态变化[23-24]。
除此之外,近期研究还发现,KIAA1429 可能是甲基转移酶复合体的新亚基,与mRNA 中m6A的形成有关[25]。在果蝇中,KIAA1429 的同源蛋白和WTAP 同源蛋白在选择性剪切过程中存在相互作用[39-40]。在人A549 细胞中,敲低KIAA1429 导致m6A 修饰水平的降低,其降低幅度大于METTL3和METTL14 敲低后的m6A 水平变化,这一结果表明,KIAA1429 对RNA 甲基化可能发挥着更加重要的作用[25,41]。
1.3.2 m6A去甲基化酶(消码器)
目前已经鉴定了两个m6A 去甲基化酶——FTO和ALKBH5,催化m6A 修饰去甲基化。FTO 作为人源ALKB 双加氧酶蛋白家族成员,最初被发现于一种融合脚趾突变小鼠体内,后被证实与人的肥胖和能量稳态有关[42],是大肠杆菌ALKB 的同源蛋白[43-44]。FTO 广泛存在于小鼠的多个组织,其中脑
组织中的含量尤为突出[45]。FTO敲除的小鼠比正常的小鼠有更高的致死率,表现为生长缓慢和体型瘦小。FTO 去甲基化酶作用的底物之一是多聚腺苷酸RNA 上的m6A[26,46]。FTO 依赖2 价铁离子和2-酮戊二酸催化m6A 去甲基化的过程要经历复杂的中间反应步骤:首先催化m6A 形成hm6A, 然后催化
hm6A 形成f6A,最后催化f6A 形成A,每一步反应都很迅速[47]。相较于ALKB 家族的其他分子,FTO的C 端有一个折叠,该结构可能通过促进蛋白质-蛋白质或蛋白质-RNA 的相互作用调节FTO 的功能。除此之外,免疫荧光实验显示,FTO部分定位在核内亚细胞器——核小斑[26,48],并通过调控m6A水平影响到pre-mRNA 加工剪切因子SRSF2 的结合,影响pre-mRNA 剪接成熟的过程[49]。近期有研究发现,甲氯芬那酸(meclofenamic acid) 可以和FTO竞争性地结合发生m6A 修饰的核酸分子,从而抑制FTO 的去甲基化作用[50]。
ALKBH5是人的大肠杆菌ALKB 双加氧酶家族同源蛋白的另一成员,是继FTO之后第二个被发现的m6A 去甲基化酶,能够分别在体外和体内去除m6A 的甲基基团[27,51]。与FTO 不同的是,ALKBH5 可直接催化m6A 到A,目前尚未发现中间产物。有研究显示,ALKBH5 倾向于结合特异的m6A 修饰的单链RNA 去甲基化[52]。本实验室利用RNA 原位杂交技术(poly(A)-tailed RNA FISH) 鉴定发现,ALKBH5 基因敲除能促进mRNA 出核,证实m6A 可能参与到mRNA 的出核转运[27]。ALKBH5定位于细胞核内亚细胞器——核小斑,且对RNA酶A 敏感,说明其作用依赖于RNA。在ALKBH5基因敲除的雄鼠模型中,mRNA 中m6A 的水平显著增加。ALKBH5 敲除还与精子发育密切相关,减数分裂中期的精母细胞发生凋亡导致精子发育受损、精子畸形,睾丸形状异常、重量变小。在ALKBH5敲除的雄鼠睾丸样品中,与正常组相比较差异表达的基因涉及多种不同的生物学功能,并包括涉及到p53 功能互作网络的精子发育相关的功能[27]。
FTO 和ALKBH5 是目前已知的m6A 去甲基化酶,其他的去甲基化酶还有待发现。由于组织特异性分布和RNA 底物不同,FTO 和ALKBH5 在RNA 的加工代谢过程中起到了不同的作用,同时,它们的基因敲除小鼠所对应的表型也不同[53]。因此,可能存在细胞和组织特异性m6A 去甲基化酶。
1.3.3 m6A结合蛋白(读码器)
m6A 广泛存在于生物体中并发挥着重要的生物学功能,其修饰水平受到甲基转移酶和去甲基化酶活性的动态调控。m6A 修饰的RNA 序列结构倾向于保持单链状态[54],可能与其结合蛋白的识别及协同作用相关。截至目前,哺乳动物中已经有一些m6A 结合蛋白被预测和发现,其中部分已经被证实了具有能够介导m6A 发挥重要调控作用的生物学功能。目前发现的m6A 结合蛋白主要是含有YTH 结构域的蛋白家族[55-56],分别是定位在细胞质的YTHDF1、YTHDF2 和定位于细胞核中的YTHDC1[8-9,15,57-59]。此外,在哺乳动物细胞中发现与m6A 关联的蛋白还有HuR 和HNRNP 等[15]。
YTHDF2 相对于YTHDF1 而言,对m6A 有更强的结合能力。PAR-CLIP 测序结果表明,该蛋白主要结合mRNA 和一些长非编码RNA,结合位点主要是在富含GAC 序列的3′ UTR 区域,和m6A 修饰区域有很大程度的重合[9]。在细胞中,YTHDF2可能与介导mRNA 翻译的核糖体RNA 竞争性地结合甲基化的转录本,进而影响RNA 的半衰期,加速RNA 的降解,影响mRNA 的稳定性[9,29]。YTHDF2的C 端可以特异性识别并结合m6A,N 端则负责将已结合的复合体引导并定位到介导RNA 降解的细胞质亚细胞器P-body[9,60]。除YTHDF2 外,其家族的另外两个同源蛋白YTHDF1和YTHDF3有与YTHDF2相似的RNA结合亚基[9]。YTHDF1已被证实与蛋白质翻译有关。人类的YTHDF1 结合m6A 修饰的mRNA 后,与起始翻译的因子eIF 及核糖体相互作
用,核糖体结合到mRNA 上及在其上滑动的速度加快,增强了这些mRNA 的翻译效率[57]。根据YTHDF1 和YTHDF2 的功能推测,YTHDF3 可能会与细胞内RNA 的代谢有关系,但是其具体功能尚不明确。YTH 家族另外一个定位于细胞质中的蛋白YTHDC2 相对分子质量很大,目前尚未被鉴定为m6A 的结合蛋白,对其功能还有待进一步的研究。
YTHDC1 定位在细胞核中的YT-body,与定位在细胞质中的YTHDF2 功能不同。PAR-CLIP 测序数据表明YTHDC1 能结合GGAC,与m6A 修饰的保守基序GGACU一致,并且其结合位点大部分位于终止密码子附近,这也与m6A的富集位点一致[58]。YTHDC1可以在核小斑处形成特定的结构,能与mRNA 剪切因子相互作用调控mRNA 剪切[59,61]。YTHDC1 与SRSF3 结合,能够促进有m6A 修饰的外显子在剪切过程中被保留。若m6A 修饰的外显子被SRSF10 结合或没有m6A 修饰的外显子被SRSF10 结合,则促进此外显子被切除。SRSF3 和SRSF10 竞争性结合YTHDC1[59]。
1.4 m6A的生物学功能
m6A 广泛存在于各种真核生物的mRNA 以及其他非编码RNA 中,但是其确切的生物学功能目前尚不明确,可能与RNA 的转录、加工、转运、翻译、降解,细胞发育分化以及细胞分裂等多个过程相关[41,62]。在哺乳动物中,m6A 动态变化可能通过调节多种与RNA 相关的细胞信号通路影响基因表达和细胞命运。
1.4.1 m6A调控mRNA加工代谢及其生物学功能 (图1B)
在拟南芥中,MTA (METTL3 同源基因) 缺陷导致胚芽发育和生长停滞[34]。在果蝇中,敲除IME4 (METTL3 同源基因) 会导致胚胎死亡[32]。酵母中m6A 调控细胞减数分裂[8]。m6A 还影响干细胞定向分化及细胞重编程和生物钟节律[19-20,63-64]。HeLa细胞中WTAP 和METTL3 基因缺陷均会导致细胞凋亡增加,而WTAP 近期被发现是急性髓性白血病发生的新致癌基因[65]。ALKBH5 基因敲除小鼠表现为精子发育异常[27],FTO 则是肥胖和2 型糖尿病相关基因[26]。YTHDF2 结合的mRNA 倾向于快速降解[9],且其基因变异与髓性白血病和急性髓性白血病发生相关[66]。此外,在胚胎干细胞中,m6A可以影响mRNA 的代谢,维持细胞的自我更新,作为转录组水平标志控制胚胎细胞的分化和细胞命运[67-68]。综上,mRNA m6A 修饰具有重要生物学功能,并和疾病的发生发展紧密相关。
mRNA m6A 修饰的甲基化和去甲基化过程主要发生在细胞核。前体pre-mRNA 的内含子区域也存在m6A,提示该甲基化修饰可能调控mRNA 前体
选择性剪切加工形成成熟的mRNA[41]。METTL3 基因沉默引起的m6A 水平下降会导致mRNA 选择性剪切发生变化,并显著富集于p53 信号通路和细胞
凋亡通路中,这种调控作用在脑组织中更为明显[16];METTL3 和拟南芥中同源基因MTA 与剪接调控蛋白WTAP (AtFIP37) 相互作用, 提示m6A 可能与mRNA剪接相关[69]。WTAP、METTL3、ALKBH5基因敲低细胞中,mRNA 表达水平和选择性剪接模式发生显著变化[24,27]。FTO 基因敲低会影响甲基化的修饰水平,进而通过影响剪切因子SRSF2 结合到mRNA 上的水平,最终导致选择性剪接成不同的转录本[41,49]。而分布在内含子区域的m6A 修饰可能调控了hnRNPC 剪接因子结合mRNA 内含子序列, 从而影响mRNA 的剪接[70]。总之,mRNAm6A修饰的RRACH 序列可能类似于顺式调控因子,调控mRNA 加工和代谢。
m6A修饰在mRNA的3′UTR区域高度富集,同时,这一区域也是microRNA 的靶向结合区域,因此,m6A 修饰可能会参与调控microRNA 与mRNA 的结合过程。在3′UTR 存在m6A 修饰的转录本中,约67% 含有至少一个microRNA 结合位点[16]。本实验室研究发现了microRNA 调控mRNA m6A 甲基化位点生成的选择性机制,同时,发现了m6A 调控细胞重编程的重要功能。microRNA 通过序列配对机制调控mRNA m6A 的水平。人为改变microRNA的表达或者突变其靶标mRNA 配对种子区的序列,可以调控与m6A 甲基转移酶亚基METTL3 结合的RNA ( 同时这些RNA 也是microRNA 靶标),进而调节m6A 的修饰水平。过表达RNA m6A 甲基转移酶催化亚基METTL3 引起的m6A 水平升高能显著提高小鼠成纤维细胞重编程为多能性干细胞的效率,反之则抑制重编程,并且使用抑制m6A 形成的小分子抑制剂环亮氨酸处理得到了和敲低METTL3一致的效果,显著降低了小鼠成纤维细胞的重编程效率[19]。
1.4.2 m6A调控非编码RNA加工代谢(图2)
通过对已有的m6A 数据进行整合分析,发现在很多长非编码RNA 上同样存在着m6A 修饰,如7SK、XIST、HOTAIR、HOTTIP、TUG1 等。从基因类型上讲,m6A 修饰主要存在于lincRNA 和antisense两种长链非编码RNA。不同于mRNA 的是,m6A在内含子区域的分布数量高于外显子区域,但m6A修饰同样倾向于富含GAC 的保守序列。通过对人源m6A 修饰的非编码基因的功能注释,发现这些基因主要参与蛋白质或者核酸的结合,表明非编码RNA 可能通过这种m6A 修饰来引导蛋白质在细胞内正确定位并结合DNA 或者RNA,进而影响转录或翻译等过程。最近有研究表明,m6A 去甲基化酶FTO可能调控了非编码microRNA 的m6A 修饰[71]。与mRNA 类似,lncRNA 中的m6A 修饰可能同样会影响多外显子的非编码RNA 选择性剪切,形成不同的转录本,进而影响到其下游功能。
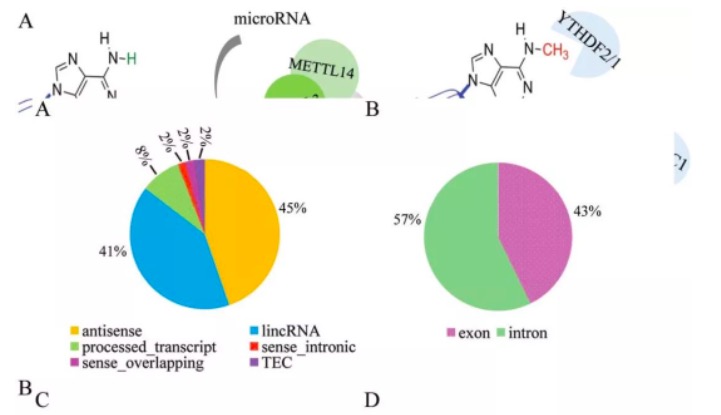
A:含有m6A修饰的非编码RNA基因类型注释。以GENCODE数据库为参考,m6A甲基化修饰的长非编码RNA主要是antisenseRNA 以及lincRNA。B:m6A修饰普遍分布于非编码RNA的编码区(外显子exon)和非编码区(内含子intron),且内含子中的分布多于外显子。C:非编码RNA m6A修饰具有与mRNA一致的保守序列:GGACU。D:m6A修饰的非编码RNA的功能富集。主要涉及:核酸结合、催化活性、蛋白质结合和离子结合。
图2 非编码RNA中m6A修饰的分布及功能富集
2 5-甲基胞嘧啶(m5C)
2.1 检测技术
除m6A 外,m5C 是另一种在RNA 中广泛存在的甲基化修饰形式。目前,DNA 中的m5C 修饰已经成为表观遗传学的研究热点,而在RNA 中的研
究则处于起步阶段,其修饰位点的分布特征以及生物学功能等仍不清楚[72]。近期,在RNA m5C 的基因组水平上的单位点检测及生物学功能方面才有初步探索[73]。
通常可以利用质谱技术检测RNA 整体水平上的m5C 修饰程度。至今为止,已有4 种方法应用于m5C 的单位点鉴定,分别为RNA 亚硫酸盐测序、m5C-RIP、Aza-IP、miCLIP[10-12]。其中RNA 亚硫酸盐测序是目前最为理想的方法,该方法可以检测到RNA 经亚硫酸盐处理后转换为尿嘧啶的非甲基化胞嘧啶,而甲基化胞嘧啶经处理后不发生转换。与亚硫酸盐测序方法不同,m5C-RIP、Aza-IP 和miCLIP方法并不依赖于化学碱基的转化,因此,可以避免过激的化学伤害造成的RNA 降解,并能在免疫沉淀的过程中富集RNA 底物,使低丰度的RNA 在普通的深度测序中同样可以被检测到,但这些方法的敏感性低于RNA 亚硫酸盐测序方法,检测到的m5C 修饰位点数量远远低于后者。早期研究受到检测技术的限制,在mRNA中仅发现少数的m5C修饰,而近期通过亚硫酸盐测序方法发现,在HeLa 细胞中mRNA 和非编码RNA 的m5C 均存在着广泛的修饰,并且得到了单位点的m5C 谱[10]。
2.2 分布特征
很多古细菌以及真核生物的tRNA 中都已经被证实存在m5C 修饰[74],修饰位点主要集中在可变臂和反密码环。有研究指出,这种修饰可以稳定tRNA 的二级结构,影响氨酰化的形成及密码子的识别[75-80]。此外,m5C 修饰还存在于rRNA 结合tRNA 发挥翻译活性的区域[81]。
2012 年,Squires 等[10] 通过亚硫酸盐处理转录组结合测序技术得到了mRNA 上的m5C 修饰位点,并指出mRNA 中的m5C 修饰主要富集在非翻译区(3′UTR 和5′UTR) 以及AGO 蛋白结合位点附近。
2.3 修饰酶(图3)
2.3.1 m5C甲基转移酶
和m6A 一样,RNA m5C 修饰也可能是动态可逆的,甲基转移酶以S- 腺苷甲硫氨酸(SAM)为供体,将甲基转移到胞嘧啶C 形成5- 甲基胞嘧啶m5C[82]。RsmB 是第一个被发现的m5C 甲基转移酶,主要催化细菌rRNA 上的甲基化形成[83]。随后,30 多种RNA 上的m5C 甲基转移酶陆续被发现,这些甲基转移酶主要可分为NOP2/NOL1、YebU/Trm4、RsmB/
Yn1022c 和PH1991/NSUN 四类[82],而且这些酶在真核生物中有很高的保守性。近年来NSUN 蛋白家族被广泛深入研究。人的NSUN 蛋白家族共有9 个蛋白[10,84],该家族的多个成员都具有潜在的m5C 甲基转移酶功能结构域,其中NSUN2 的催化活性已被证实[85]。
RNA受到甲基转移酶,如NSUN家族蛋白催化C形成甲基化的m5C修饰,而去甲基化酶,如可能性较大的TET家族蛋白催化其去甲基化。
图3 m5C修饰的动态可逆过程
NSun2 和Dnmt2 被认为可能是哺乳动物的RNA m5C 甲基转移酶[10-11],它们的催化位点有一定的交集。其他的酶,如NSUN1 和NSUN3~7 被预测可以催化一些保守的甲基化位点的甲基化过程[86]。NSUN1、NSUN2 和NSUN5 已被证实能结合RNA,但这些酶的特异性结合底物并不是很清楚[87]。NSUN蛋白家族的生物学功能至今还不是非常清楚,但是它们都在小鼠的胚胎发育过程中有表达,NSUN2~7的转录本富集在发育的脑中并对神经发育有重要调控作用[86]。小鼠的NSUN2 是染色质体的重要组成成分,而且对于表皮干细胞的自我更新和分化有密切的联系[88]。NSUN2 能够催化tRNA的甲基化,而且在mRNA 和非编码RNA 中也有一定作用[10-12]。另外, 已有研究表明,DNMT2 是tRNA 的甲基转移酶[89]。目前发现的多数的甲基转移酶定位在细胞核内,表明这些酶本身以及其调控的甲基化过程可能对RNA 的加工有作用。NSUN2最初在哺乳动物上皮中被发现作为原癌基因c-Myc的转录靶点,且NSUN2 在多种癌组织中呈上调趋势,人鳞状细胞癌中NSUN2 基因敲低抑制了其生长能力。NSUN2 还是能被AuraroB 激酶调控的核仁蛋白,在肿瘤细胞中通过稳定分裂期纺锤体而促进细胞分裂。然而,这一功能似乎与其甲基转移酶功能无关,且有待被体内实验验证。
此外,NSUN2 敲除小鼠呈现出与DNMT2 敲除斑马鱼类似的表型,NSUN2 敲除小鼠体型偏小,且在包括皮肤和睾丸等的特异性组织的后期发育受到延迟或阻碍,因此,NSUN2 可以维持鼠表皮细胞的正常分化[88]。在人中,NSUN2 的几个遗传突变已被鉴定,且初步导致常染色体隐性的智障以及Dubowitz 样综合征,其共同特征为生长和心智阻滞,面部异常以及皮肤畸形[90]。RNA 甲基化缺失是否是这些复杂疾病的诱因以及如何致病尚不清楚。然而,与人的综合征相似的是,果蝇中NSUN2 同源基因敲除导致严重的短期记忆异常,而DNMT2 和NSUN2 双敲除使得所有的tRNA 甲基化丢失,特异性影响脑、肝脏和脂肪组织的发育[91]。
NSUN1 (NOP2) 是核仁蛋白,能够结合60~80S的核糖体前体颗粒,目前已知其主要功能在于调控细胞分裂[92]。NSUN1 能否甲基化核糖体RNA 还有待被证实。此外,NSUN1 基因定位于具有智能缺陷和小头畸形等特征的Cri-du-chat 综合征患者中缺失的基因组区域。NSUN4 主要作用于线粒体中由线粒体DNA 编码的12S rRNA 的C911 位点。与NSUN2 基因敲除不同的是,生殖细胞中NSUN4 基因敲除可致死,8.5 d 的胚胎表现出严重的生长迟钝并缺乏明显的可辨别解剖学结构[93]。心脏中的NSUN4 条件性敲除可诱发心肌病和因线粒体核糖体组装受损和线粒体翻译受抑制后导致的呼吸链缺陷。
虽然酵母的NSUN5同源基因Rcm1已被报道能特异性修饰25S rRNA[94], 然而, 截至目前NSUN5的生物学功能和RNA 底物尚未可知。在人中,NSUN5 基因定位于在Williams-Beuren 综合征患者中缺失的基因组区域,这是一种罕见的神经发育紊乱的疾病,缺乏NSUN5 可能与该疾病的生长迟缓、肌肉病变,或早老等表型相关。NSUN7 基因突变可导致精子的运动能力受损,可能与人和小鼠的不育症相关[95]。
NSUN3和NSUN6蛋白的生物学功能尚属未知。总之,目前对于RNA m5C 甲基转移酶的生物学功能的证据非常有限,然而一些共性却已凸显。首先,NSUN 蛋白多与人类综合性疾病相关,包括生长迟缓和神经发育缺陷等。rRNA 和tRNA 的m5C 修饰能直接调控总的蛋白质翻译,或许能够解释这一关联。而许多缺乏RNA 甲基转移酶的器官之所以呈现变小的表型,可能是因为蛋白质合成途径可关联到细胞的大小。另外,一个共性在于RNA甲基转移酶缺陷对脑和睾丸的影响较为普遍,而这两者也是对蛋白质翻译速率变化反应最为敏感的器官。
2.3.2 m5C去甲基化酶
Tet 家族蛋白依赖2 价铁离子和2- 酮戊二酸催化DNA m5C 去甲基化过程,但在RNA 中还未发现能够去除m5C 甲基化的酶,此前有报道称RNAm5C 的去甲基化过程也可能是由Tet 催化完成,但仍需要实验进一步验证[96]。
2.4 m5C的生物学功能
2.4.1 m5C在mRNA中的功能
相较于m6A,m5C 在mRNA 中的功能研究还并不深入。基于现有的一些研究基础,我们推测,m5C 的修饰可能会调控mRNA 的选择性剪切,m5C修饰水平可能会影响外显子的保留水平以及转录本的组装形式。除此之外,m5C 在RNA 翻译起始位点的富集提示其可能与蛋白质翻译有较为密切的关系[97]。而且,m5C 可能和mRNA 的稳定性有关:含有m5C 修饰的 mRNA 其稳定性较强,而3′UTRm5C 水平的降低可以导致mRNA 的稳定性降低[97]。鉴于mRNA 中m5C 的修饰位点富集在AGO 蛋白的结合位点附近,m5C 修饰可能会影响microRNA 参与的RNA 降解途径, 但miCLIP 方法表明在NSUN2 缺失的情况下,其靶标mRNA 的表达并不会有明显降低,因此,目前并没有足够的证据证明m5C 是否可以调控mRNA 的稳定性[98]。NSUN2 基因的突变会引起常染色体隐性综合征,表现为智力障碍、皮肤问题和生长迟缓[90,99-101]。总之,m5C 修饰在RNA 中广泛存在,而且其功能可能涉及到细胞内信号转导、组织发育分化和癌症等许多方面[102-103]。
2.4.2 m5C在非编码RNA中的功能
多项研究表明,m5C 在tRNA 和rRNA 中有重要的作用[104-106]。在tRNA 中,m5C 主要存在于可变区和反密码子环,从而使tRNA 的二级结构更加稳定,增强密码子识别的能力[75]。在DNMT2 和NSUN2 双敲除小鼠中,tRNA 中的m5C 修饰消失,tRNA 的功能减弱从而抑制蛋白质的合成[107]。在其他物种,如酵母、果蝇、斑马鱼中,tRNA m5C 和蛋白质的翻译和组织分化有关[108-110],而发生在rRNA 中的m5C 修饰被认为和翻译过程有关[81]。
在长非编码RNA (lincRNA),如HOTAIR 和XIST 中,m5C 修饰存在于lincRNA 和染色质相互结合作用的功能区,这些转录后的修饰增加了长非编码RNA 结构的多样性。有研究表明,Nsun2 催化的m5C 的修饰会发生在穹窿体RNA 上(Vault RNA),这是一种存在于穹窿体核糖核蛋白复合物中的非编码RNA。这种修饰能够调控自身加工成特异的小RNA[12]。
3 展望
尽管RNA 修饰早已被发现,但其具体功能还知之甚少。这些修饰对个体生命活动的影响将会是以后RNA 领域研究的重点与热点。
目前,在m6A 修饰领域中,研究者已发现其甲基转移酶、去甲基化酶及结合蛋白等较为完整的酶学系统。microRNA 等微调控的机制也已越来越清楚,但由于目前实验和测序技术限制,mRNAm6A 单位点鉴定仍是很大的挑战。与之相比,mRNA m5C 利用亚硫酸盐测序能够较精确地定位m5C 单位点,但相关的酶系统基本还是空白,需要尽快完善补充。与m6A 类似,m5C 在3′UTR 也有明显的富集,且与AGO 蛋白引导的microRNA 靶标mRNA 3′UTR 序列有重合,预示microRNA 对m5C 可能也有微调作用。m6A 和m5C 在pre-mRNA内含子区也有富集,对mRNA 的选择性剪接可能发挥着一定的作用,需要进一步深入研究。
m6A 的RNA 甲基转移酶可能是一个包含多个亚基的酶复合体,目前只发现约180 kDa 的METTL3/METTL14/WTAP 三聚体,因此,通过免疫共沉淀和同源搜索等方法找到并确定更多的m6A 甲基转移酶和去甲基化酶,可以更清晰地阐述m6A 甲基化形成的选择性机制。另外,还需要找到更多的m6A 结合蛋白,这些蛋白可以通过结合甲基化位点,从而招募其他的重要分子,如剪切因子等结合到RNA上进而发挥相应的功能。但是到目前为止,已经确
定能够结合m6A 并行使功能的m6A 结合蛋白只有YTHDF2、YTHDF1 和YTHDC1。因此,该领域的研究是扩展m6A 修饰功能的重要突破口。除了
m6A,对于其他的修饰形式,如m5C,其相应的甲基转移酶和去甲基化酶的发现对研究其机制和功能均起着重要的推进作用。
m6A 可能会影响细胞内的多项生命活动,如mRNA 转录、选择性剪切、出核转运、翻译、降解等过程,但目前已被证实的只有YTHDF2 介导的mRNA 降解、YTHDF1 介导的mRNA 翻译以及YTHDC1 介导的选择性剪切,其他的生物学功能亟待去揭示。而对于m5C 修饰的功能,目前也知之甚少。因此,深入了解m6A 和m5C 修饰的功能是表观转录组研究的一项重要任务。
除mRNA 外,m6A 和m5C 在非编码RNA 中也存在,非编码RNA 在加工成熟过程中也经历了选择性剪接、形成高级结构等复杂的过程。这些甲基化修饰在非编码RNA 中很可能会同影响mRNA一样影响非编码RNA 的选择性剪接。
除上述m6A 和m5C 外,RNA 还存在其他约150 余种化学修饰,这些修饰必定对RNA 的功能起到了不容忽视的作用。随着技术手段的不断提高,期待这些修饰在不久的将来能得到透彻地研究,进而对生命活动现象的理解和疾病的诊治提供理论基础。
[参 考 文 献]
[1] Cantara WA, Crain PF, Rozenski J, et al. The RNA
modification database, RNAMDB: 2011 update. Nucleic
Acids Res, 2011, 39: D195-201
[2] Beemon K, Keith J. Localization of N6-methyladenosine
in the Rous sarcoma virus genome. J Mol Biol, 1977, 113:
165-79
[3] Krug RM, Morgan MA, Shatkin AJ. Influenza viral
mRNA contains internal N6-methyladenosine and
5′-terminal 7-methylguanosine in cap structures. J Virol,
1976, 20: 45-53
[4] Rottman FM, Desrosiers RC, Friderici K. Nucleotide
methylation patterns in eukaryotic mRNA. Prog Nucleic
Acid Res Mol Biol, 1976, 19: 21-38
[5] Schibler U, Kelley DE, Perry RP. Comparison of
methylated sequences in messenger RNA and
heterogeneous nuclear RNA from mouse L cells. J Mol
Biol, 1977, 115: 695-714
[6] Chen-Kiang S, NevinsJR, Darnell J. N-6-methyladenosine
in adenovirus type 2 nuclear RNA is conserved
in the formation of messenger RNA. J Mol Biol, 1979,
135: 733-52
[7] Wei CM, Moss B. Nucleotide sequences at the N6-
methyladenosine sites of HeLa cell messenger ribonucleic
acid. Biochemistry, 1977, 16: 1672-6
[8] Schwartz S, Agarwala SD, Mumbach MR, et al. Highresolution
mapping reveals a conserved, widespread,
dynamic mRNA methylation program in yeast meiosis.
Cell, 2013, 155: 1409-21
[9] Wang X, Lu Z, Gomez A, et al. N6-methyladenosinedependent
regulation of messenger RNA stability. Nature,
2014, 505: 117-20
[10] Squires JE, Patel HR, Nousch M, et al. Widespread
occurrence of 5-methylcytosine in human coding and noncoding
RNA. Nucleic Acids Res, 2012, 40: 5023-33
[11] Khoddami V, Cairns BR. Identification of direct targets
and modified bases of RNA cytosine methyltransferases.
Nat Biotechnol, 2013, 31: 458-64
[12] Hussain S, Sajini AA, Blanco S, et al. NSun2-mediated
cytosine-5 methylation of vault noncoding RNA
determines its processing into regulatory small RNAs.
Cell Rep, 2013, 4: 255-61
[13] Perry RP, Kelley DE, Friderici K, et al. The methylated
constituents of L cell messenger RNA: evidence for an
unusual cluster at the 5′ terminus. Cell, 1975, 4: 387-94
[14] Wei CM, Gershowitz A, Moss B. Methylated nucleotides
block 5′ terminus of HeLa cell messenger RNA. Cell,
1975, 4: 379-86
[15] Dominissini D, Moshitch-Moshkovitz S, Schwartz S, et
al. Topology of the human and mouse m6A RNA
methylomes revealed by m6A-seq. Nature, 2012, 485:
201-6
[16] Meyer KD, Saletore Y, Zumbo P, et al. Comprehensive
analysis of mRNA methylation reveals enrichment in 3′
UTRs and near stop codons. Cell, 2012, 149: 1635-46
[17] Golovina AY, Dzama MM, Petriukov KS, et al. Method
for site-specific detection of m6A nucleoside presence in
RNA based on high-resolution melting (HRM) analysis.
Nucleic Acids Res, 2014, 42: e27
[18] Liu N, Parisien M, Dai Q, et al. Probing N6-methyladenosine
RNA modification status at single nucleotide
resolution in mRNA and long noncoding RNA. RNA, 2013, 19: 1848-56
[19] Chen T, Hao YJ, Zhang Y, et al. m6A RNA methylation is
regulated by microRNAs and promotes reprogramming to
pluripotency. Cell Stem Cell, 2015, 16: 289-301
[20] Wang Y, Li Y, Toth JI, et al. N6-methyladenosine
modification destabilizes developmental regulators in
embryonic stem cells. Nat Cell Biol, 2014, 16: 191-8
[21] Fu Y, Dominissini D, Rechavi G, et al. Gene expression
regulation mediated through reversible mA RNA
methylation. Nat Rev Genet, 2014, 15: 293-306
[22] Lee M, Kim B, Kim VN. Emerging roles of RNA
modification: m6A and U-tail. Cell, 2014, 158: 980-7
[23] Liu J, Yue Y, Han D, et al. A METTL3-METTL14
complex mediates mammalian nuclear RNA N6-adenosine
methylation. Nat Chem Biol, 2014, 10: 93-5
[24] Ping XL, Sun BF, Wang L, et al. Mammalian WTAP is a
regulatory subunit of the RNA N6-methyladenosine
methyltransferase. Cell Res, 2014, 24: 177-89
[25] Schwartz S, Mumbach MR, Jovanovic M, et al.
Perturbation of m6A writers reveals two distinct classes of
mRNA methylation at internal and 5′ sites. Cell Rep,
2014, 8: 284-96
[26] Jia G, Fu Y, Zhao X, et al. N6-methyladenosine in nuclear
RNA is a major substrate of the obesity-associated FTO.
Nat Chem Biol, 2011, 7: 885-7
[27] Zheng GQ, Dahl JA, Niu YM, et al. ALKBH5 is a
mammalian RNA demethylase that impacts RNA
metabolism and mouse fertility. Mol Cell, 2013, 49: 18-29
[28] Tuck MT. Partial purification of a 6-methyladenine mRNA
methyltransferase which modifies internal adenine
residues. Biochem J, 1992, 288: 233-40
[29] Bokar JA, Rath-Shambaugh ME, Ludwiczak R, et al.
Characterization and partial purification of mRNA N6-
adenosine methyltransferase from HeLa cell nuclei.
Internal mRNA methylation requires a multisubunit
complex. J Biol Chem, 1994, 269: 17697-704
[30] Bokar JA, Shambaugh ME, Polayes D, et al. Purification
and cDNA cloning of the AdoMet-binding subunit of the
human mRNA (N6-adenosine)-methyltransferase. RNA,
1997, 3: 1233-47
[31] Bujnicki JM, Feder M, Radlinska M, et al. Structure
prediction and phylogenetic analysis of a functionally
diverse family of proteins homologous to the MT-A70
subunit of the human mRNA: m6A methyltransferase. J
Mol Evol, 2002, 55: 431-44
[32] Clancy MJ, Shambaugh ME, Timpte CS, et al. Induction
of sporulation in Saccharomyces cerevisiae leads to the
formation of N6-methyladenosine in mRNA: a potential
mechanism for the activity of the IME4 gene. Nucleic
Acids Res, 2002, 30: 4509-18
[33] Hongay CF, Orr-Weaver TL. Drosophila inducer of
MEiosis 4 (IME4) is required for Notch signaling during
oogenesis. Proc Natl Acad Sci USA, 2011, 108: 14855-60
[34] Zhong S, Li H, Bodi Z, et al. MTA is an Arabidopsis
messenger RNA adenosine methylase and interacts with a
homolog of a sex-specific splicing factor. Plant Cell, 2008,
20: 1278-88
[35] Bodi Z, Zhong S, Mehra S, et al. Adenosine methylation
in Arabidopsis mRNA is associated with the 3′ end and
reduced levels cause developmental defects. Front Plant
Sci, 2012, 3: 48
[36] Havugimana PC, Hart GT, Nepusz T, et al. A census of
human soluble protein complexes. Cell, 2012, 150: 1068-
81
[37] Horiuchi K, Kawamura T, Iwanari H, et al. Identification
of Wilms’ tumor 1-associating protein complex and its
role in alternative splicing and the cell cycle. J Biol Chem,
2013, 288: 33292-302
[38] Little NA, Hastie ND, Davies RC. Identification of WTAP,
a novel Wilms’ tumour 1-associating protein. Hum Mol
Genet, 2000, 9: 2231-9
[39] Granadino B, Campuzano S, Sanchez L. The Drosophila
melanogaster fl(2)d gene is needed for the female-specific
splicing of Sex-lethal RNA. EMBO J, 1990, 9: 2597-602
[40] Ortega A, Niksic M, Bachi A, et al. Biochemical function
of female-lethal (2)D/Wilms’ tumor suppressor-1-
associated proteins in alternative pre-mRNA splicing. J
Biol Chem, 2003, 278: 3040-7
[41] Yang Y, Sun BF, Xiao W, et al. Dynamic m6A modification
and its emerging regulatory role in mRNA splicing. Sci
Bull, 2014, 60: 21-2

