代谢工程利用基因改造将生物体内代谢通量重新定向到目标产物合成,从而构建高效工业菌株,实现绿色生物制造。基因组尺度代谢网络模型(Genome-scale metabolic model,GEM)可以计算特定菌种中产物合成的最优途径,预测代谢工程改造的可能靶点。通量平衡分析(Flux balance analysis,FBA)是GEM分析中最广泛使用的设计算法,但使用该方法需要掌握一定的编程技能,使得广大实验生物学家难以应用这一方法进行途径和靶点设计。此外,FBA的计算结果通常是一个包含几十个反应的列表,理清这些反应的代谢流分布进而发现有趣的代谢特征是一项耗时且依赖生物学经验的工作。因此,开发一款基于web的代谢网络模型分析工具,为不善于编程的实验生物学家赋能,使其可以借助GEM分析发现可能的代谢工程改造靶点非常有意义。

根据用户途径设计时采用的底盘菌种代谢网络模型的可靠性,CAVE提供了两种工作流程。对于质量较高的代谢网络模型,用户可以直接选择模型,设定目标产物计算得到最优途径并进行可视化(如图1中绿色箭头所示),整个过程只需要在网站上选择点击,可以在一分钟之内完成,大大降低了用户基于GEM进行途径计算分析的使用门槛,提升了工作效率。在更多情况下,数据库中或文献发表的模型虽然针对生物质生成、底物利用等进行了分析优化,却并没有针对各种生物产品合成途径进行检查质控,因此计算得到的途径可能存在各种各样的错误。CAVE提供的途径可视化功能可以帮助具有相关生物学背景知识的生物学家更容易地发现途径中可能存在的错误,进而分析造成错误的可能原因或模型中的一些不合理设置。CAVE允许用户对模型中的反应方程、可逆性、边界条件及GPR(基因-蛋白-反应)关系进行修正或添加删除反应,通过计算-检查-修改的循环迭代不断修改完善模型,保证得到合理可靠的途径指导代谢改造靶点设计(如图1中绿色箭头所示)。
为了使实验生物学家更方便的使用CAVE进行代谢途径设计和分析,CAVE提供了如下特色功能:
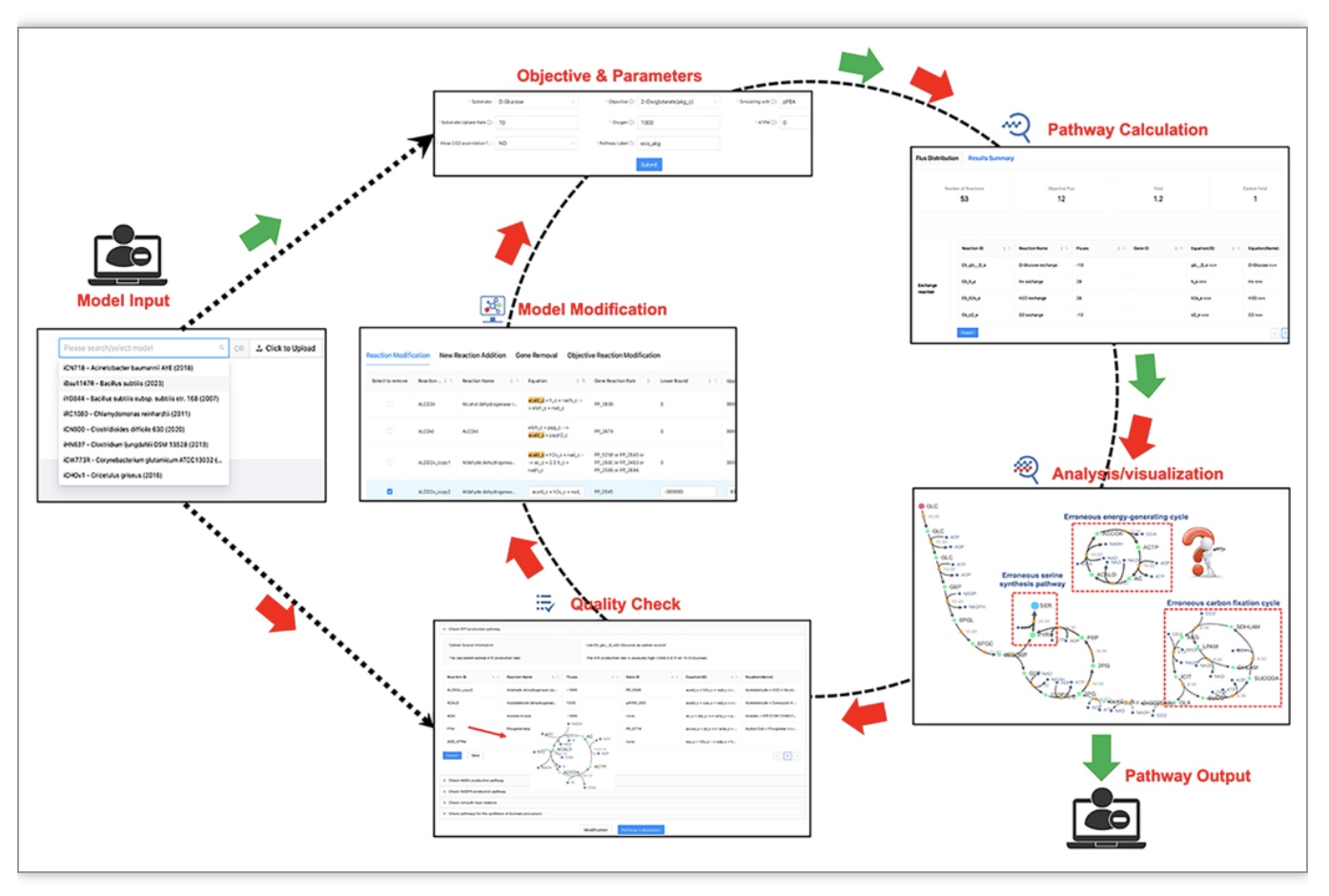
图1. CAVE分析流程
(1) CAVE内含一百多个GEM模型,并将不断更新。对于不熟悉代谢网络模型的用户,可以直接根据菌种名称选择模型进行分析而不需要自己去文献或数据库查找模型。同时, CAVE还允许用户上传自己构建或修改的模型进行分析。
(2)CAVE针对代谢途径计算中容易出现的问题,在途径计算页面引导用户设置相关参数以得到可靠结果,例如将维持速率归零、简单设置厌氧好氧等。计算结果以可搜索和可排序的智能表格形式呈现,并可根据通量大小等规则查找过滤和选择反应,还提供了统计汇总信息以快速分析途径的物料和能量平衡、产品得率等,从而更容易对途径计算结果进行分析和检查评估。
(3)CAVE依托d3flux可以直接由代谢途径的反应列表自动生成可视化途径图,并可以拖动隐藏图中节点等更容易的检查途径中的代谢流分布,以发现可能的改造靶点或检查途径中可能存在的问题。这种可视化方法摆脱了对人工绘制代谢途径图的依赖,在节省时间同时使用户可以对更多菌种代谢网络进行分析。
(4)CAVE基于云计算无服务器技术开发,可以满足大量用户高并发访问需求,并缓存用户计算得到的途径,方便用户随时随地对近期所得途径进行比较分析。
CAVE文章中提供了两个应用应用案例并在网站Tutorial部分进行了更详细的介绍。一个是通过大肠杆菌代谢模型iML1515计算α-酮戊二酸(AKG)合成途径,图2中给出了从模型选择到目标产物设定、途径计算分析和可视化的具体操作步骤。可视化的途径可以清晰的展现苏氨酸旁路(Threonine bypass)通过回收碳提高了产品的得率(图2D的途径图)。
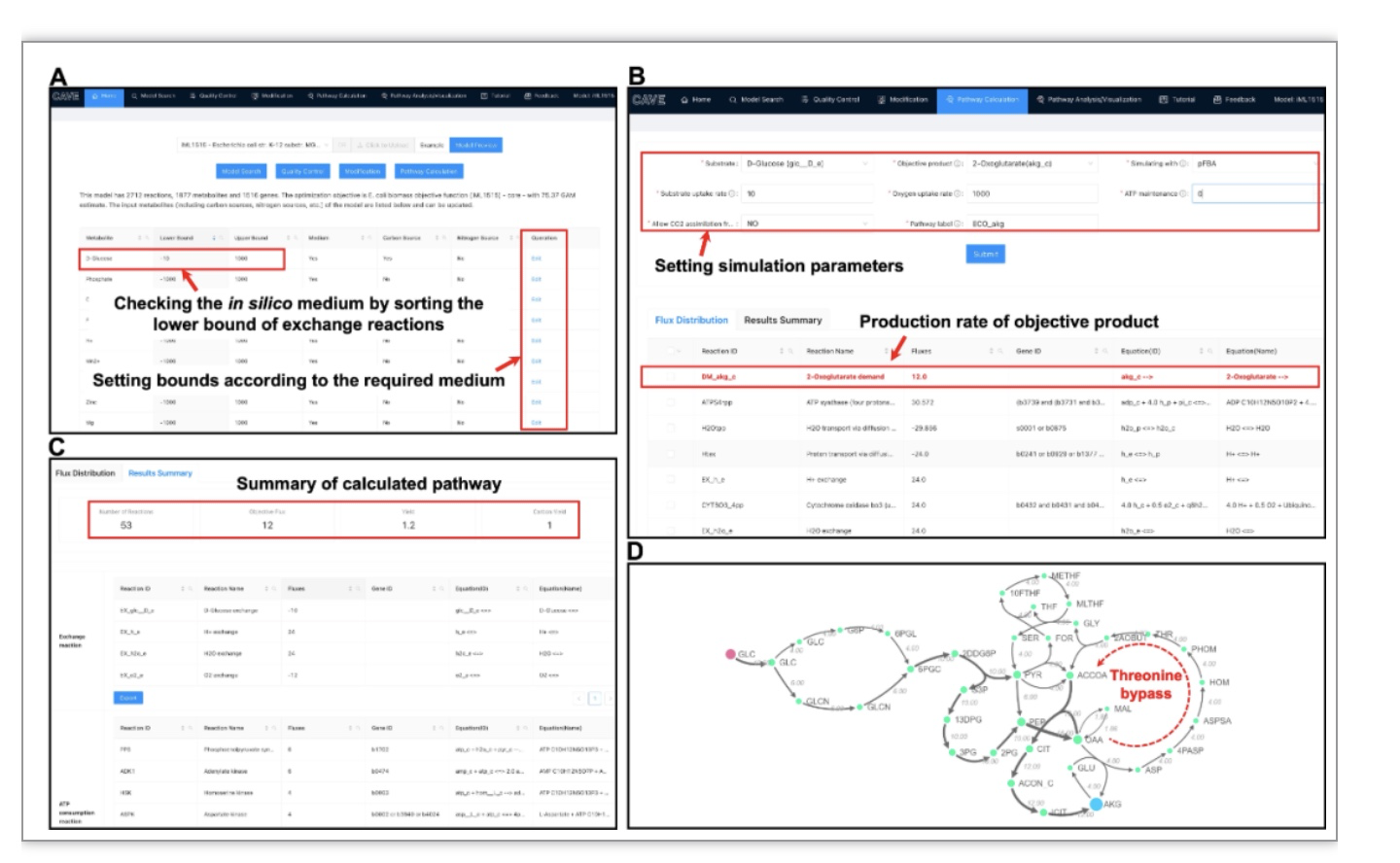
图2. iML1515模型计算AKG最优合成途径
第二个案例使用恶臭假单胞菌代谢模型iJN746来计算丝氨酸(SER)的最优合成途径,通过该案例详细说明了如何通过可视化快速发现所得代谢途径中的错误,定位错误的原因并相应对模型进行修正,如更正反应的边界条件和可逆性等,最后得到准确可靠的途径(详细步骤参加网站tutorial)。
CAVE是一个旨在帮助实验生物学家进行GEM分析及可视化的有力工具。其使生物学家无需掌握编程技能和安装软件,即可通过互联网随时随地选择模型和目标产品、计算最优途径并进行可视化分析,整个过程仅需简单选择设置和点击提交即可实现,显著降低了相关模型计算分析方法的使用门槛,提升了代谢途径和代谢改造靶点设计的工作效率。通过整合模型质量控制和修改功能,具有相关生化背景知识的生物学用户还可以通过可视化更容易地发现途径中可能存在的错误并通过迭代的计算、检查和修改过程不断提高模型质量,保证所得途径的生物学可靠性,避免不正确的途径靶点设计结果造成人力、时间和资源的浪费。总之,CAVE可以帮助生物学家快速设计、可视化和优化代谢途径,为菌种的代谢工程改造提供指导,为合成生物学研究和应用提供强大的支持。未来CAVE将整合更多的基于代谢模型分析的靶点设计算法,使生物学家可以直接得到针对目标产品的过表达、插入、弱化和敲除靶点。

