近年来,胚胎培养基(spent embryo culture media,SECM)中的游离DNA(cell-free DNA,cfDNA)引起了人们的关注,因为它提供了一种新颖的无创植入前遗传学检测(noninvasive preimplantation genetic testing,niPGT)样本来源[1, 2]。然而,SECM可能来自多种细胞类型,并导致假阴性或假阳性染色体非整倍体结果,在检测分析方法上仍然存在难点。
该文章展示了经过优化和验证的SECM中cfDNA全基因组DNA甲基化测序技术(SECM-PBAT)的实验方法和生物信息学分析流程。该技术可通过DNA甲基化信息追溯SECM中cfDNA的细胞来源,并同时检测染色体非整倍性。研究团队于2021年采用该方法揭示SECM中cfDNA的细胞来源包括囊胚细胞、卵泡颗粒细胞和极体细胞,并通过整合分析提高SECM检测非整倍体胚胎的准确性[3]。
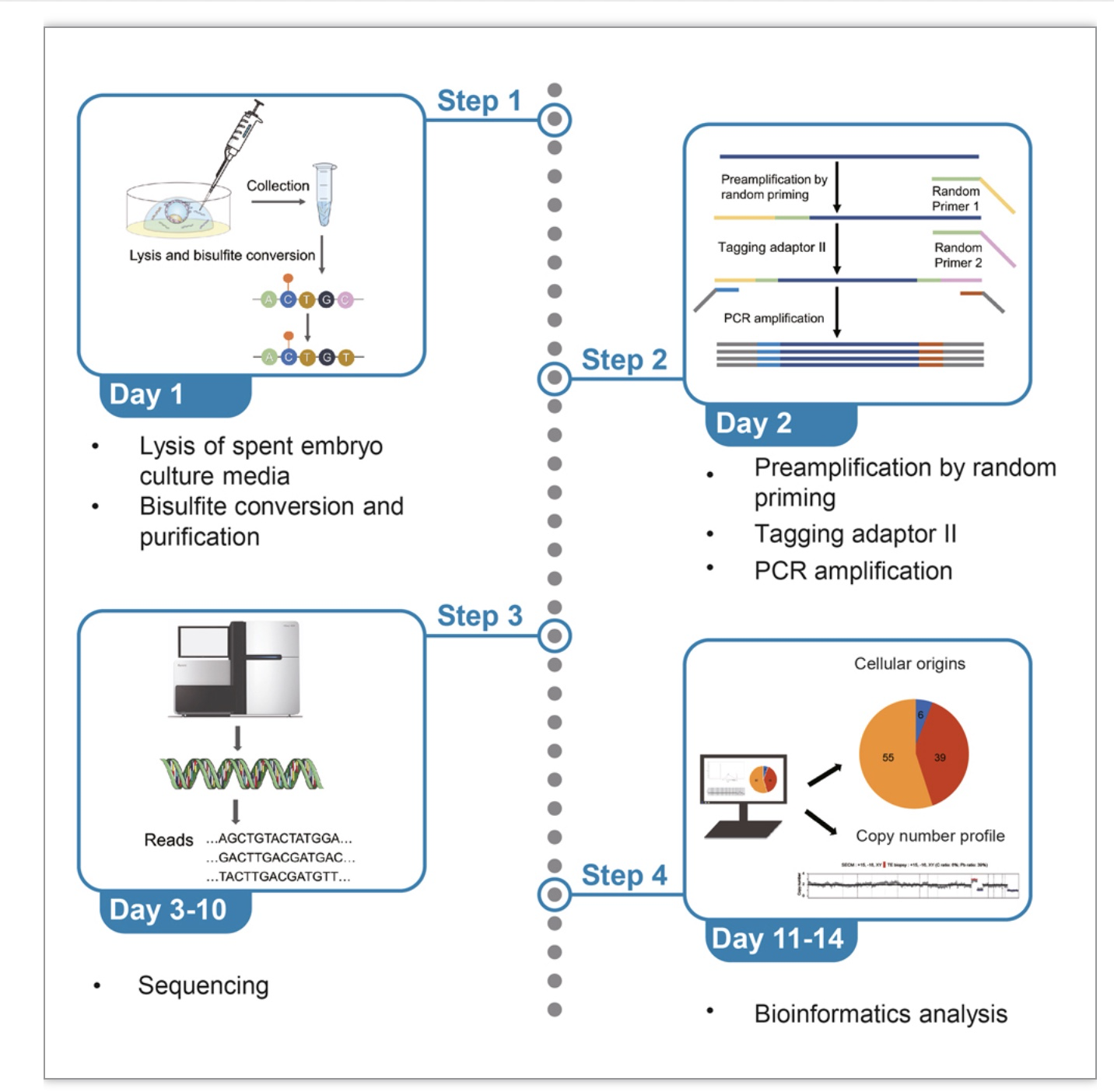
研究者详细阐述了SECM-PBAT技术的实验方法和生信分析流程(图1)。SECM-PBAT实验方法从单细胞全基因组DNA甲基化测序PBAT(Post-Bisulfite Adaptor Tagging)技术衍生而来,具体步骤包括体外胚胎培养、SECM收集、SECM裂解、亚硫酸氢盐转化、随机引物扩增、PCR扩增、文库质量控制和测序。SECM(5-20 μL)被直接裂解后进行后续步骤处理,方法高度灵敏度,可以检测低至单个细胞基因组DNA。
SECM-PBAT的生信分析流程包括数据处理、染色体非整倍性分析和细胞来源推断分析。2018年,北京大学汤富酬教授和北医三院乔杰教授团队采用单细胞PBAT技术全面绘制了人类植入前胚胎的DNA甲基化图谱[4]。以此为基础,研究者获得了769个卵母颗粒细胞和548个极体细胞特异性差异甲基化区域,并通过这些区域的DNA甲基化值反向推断SECM中的细胞来源[3]。研究团队开发了PBAT检测基因组拷贝数变异的分析方法[5]。将两种信息结合在一起,SECM-PBAT可提高SECM检测非整倍体胚胎的准确性。
图1:鉴定SECM中cfDNA来源及染色体倍性的实验和分析流程图
(图源:Gao Y et al., STAR Protoc, 2023)
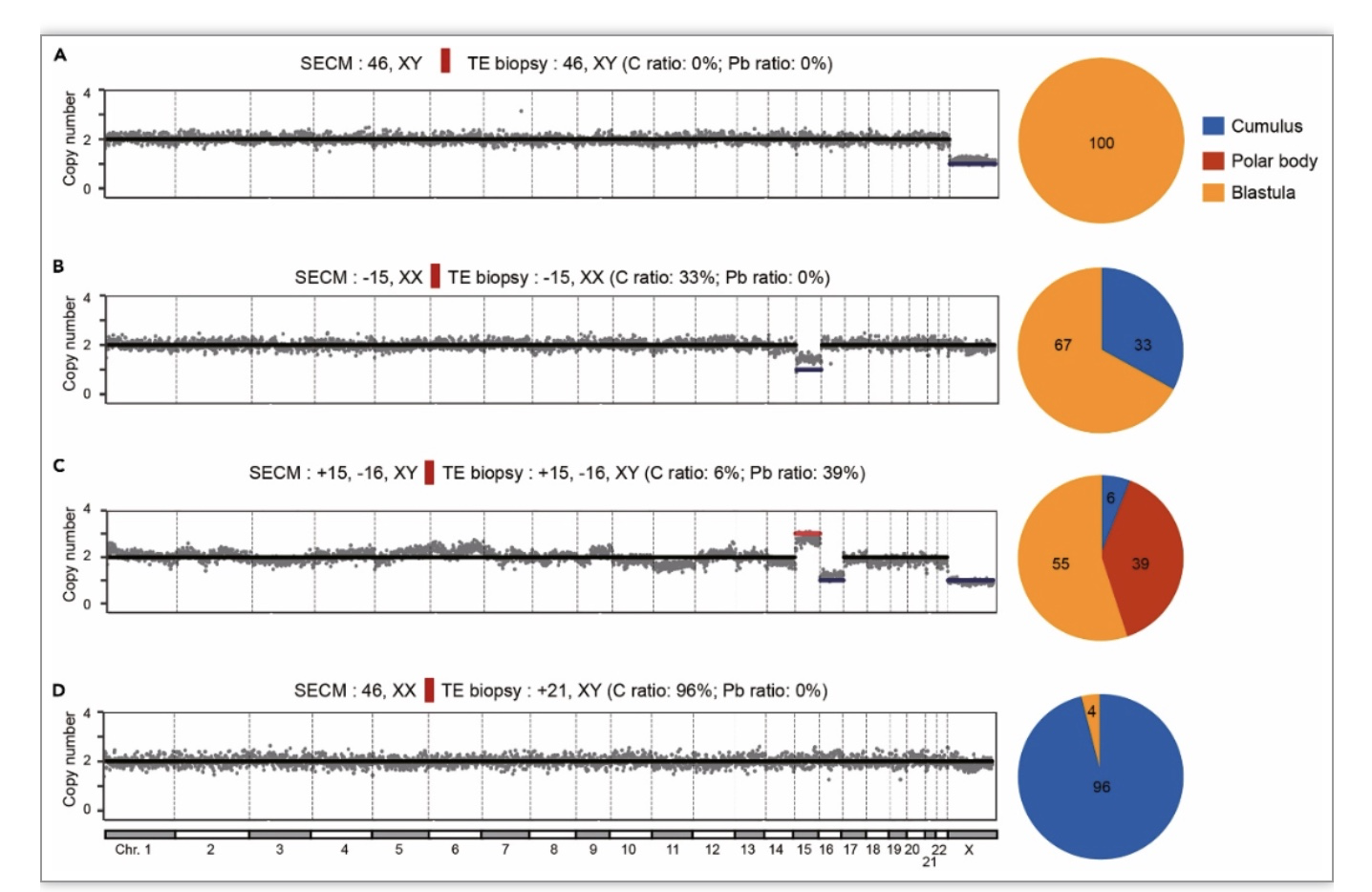
该方法对于每个样本进行了约 5-Gigabase (Gb)的测序,平均可以获得 150 万次干净读段和 12 万次唯一比对到基因组的读段,平均覆盖3万个CpG位点(≥6×)。图2显示了四个SECM cfDNA样品的细胞来源与非整倍性检测结果,图中可见SECM-PBAT技术检测出这些样品含有不同比例的囊胚细胞,颗粒细胞和极体细胞组分,并同时检测出染色体非整倍性。
图2:同时检测SECM中cfDNA染色体非整倍体及细胞起源
(图源:Gao Y et al., STAR Protoc, 2023)
综上所述,该实验方案系统性介绍了利用SECM-PBAT方法同时进行SECM中cfDNA的细胞来源推断和染色体非整倍性分析的详细流程(图1),并提供了实验过程中的注意事项与常见问题解决方案。SECM的DNA甲基化信息为人类植入前胚胎的研究和诊断提供了一个新的窗口,本方法将为该领域研究和无创植入前遗传学诊断提供重要基础。