摘 要:非编码RNA 是各种不作为翻译蛋白质的模板的RNA 的统称,参与很多生命活动的调控并扮演着重要的角色。它们参与mRNA 翻译、RNA 剪切、化学反应催化,以及参与DNA 复制修复、基因转录调控、发育和细胞分化调控等。现针对哺乳动物细胞内非编码RNA 调控依赖于RNA 聚合酶II,产物为mRNA 的转录进行综述。
关键词:非编码RNA ;基因转录;调控机制
中图分类号:Q52 文献标志码:A
Regulation of gene transcription by non-coding RNAs
LIU Xu, SHAN Ge*
( School of Life Science, University of Science and Technology of China, Hefei 230026, China )
Abstract: Non-coding RNAs (ncRNAs) are RNA molecules that do not serve as template for translation. They participate in a myriad of biological processes and play crucial regulatory roles. NcRNAs have been found to regulate translation and splicing, serve as ribozymes, modulate DNA replication and gene transcription, dictate development and cell differentiation. In this review, we focus on the regulation of ncRNAs in the transcription of RNA polymerase II in mammalian cells.
Key words: non-coding RNA; transcription; regulatory mechanism
细胞内RNA 可以分为信使RNA ( 即mRNA) 和非编码RNA (non-coding RNA,即ncRNA) 两大类。非编码RNA是各种不翻译成蛋白质的RNA的统称;但是这并不意味着这些RNA 不包含信息或者没有功能[1]。哺乳动物或者其他高等动物的大部分基因组的转录产物最终并不翻译成蛋白质,而是作为非编码RNA 存在,如人类基因组中只有2% 的区域编码mRNA,其他至少80% 的区域也发生转录,而转录产物是非编码RNA[2-3]。众所周知,细胞作为生物结构和功能的基本单位,其中发生的代谢过程、遗传信息传递等生理活动必定是受到高度调节的,那么这些不编码蛋白质的非编码RNA 在转录组中大量存在的意义是什么;它们在细胞中起什么样的作用;细胞中所有的非编码RNA 都是有功能的,还是大部分仅仅是无用的转录副产物[4-5] ;如何将这些RNA 进行分类,这些问题一直是近十年来RNA 领域的研究热点。
非编码RNA,包括在细胞中大量存在并起重要功能的RNA,如转运RNA (tRNA)、核糖体RNA(rRNA)、小核仁RNA (small nucleolar RNA, snoRNA)、微小RNA (microRNA)、小干扰RNA (small interfere RNA, siRNA)、小核RNA (small nuclear RNA, snRNA)、piRNA (Piwi-interacting RNA),多种类型的长链非编码RNA (long non-coding RNA, lncRNA) 以及环状RNA (circular RNA, circRNA) 等[6-7]。根据以往的研究,大量非编码RNA 参与了很多生命活动的调控并扮演着重要的角色,包括参与翻译过程、催化RNA 前体中内含子的剪切、DNA 复制、基因的转录调控、调控细胞发育和分化等[1,7-9]。
生物遗传信息传递的第一步是DNA 到RNA的信息传递,即转录,转录过程直接或间接影响基因的表达以及生命活动。非编码RNA 对基因表达的调控中最重要的就是对基因转录的调控[10-11]。本文将针对哺乳动物细胞内调控基因转录( 依赖RNA聚合酶Ⅱ,产物为mRNA 的转录) 的非编码RNA进行总结和讨论。
1 小分子RNA对基因转录的调控
小RNA 主要有我们熟知的microRNA、siRNA、piRNA,以及一大类启动子附近产生的RNA,包括转录起始小RNA (tiRNAs)[12]、启动子相关小RNA (promoter-associated small RNAs, PASRs) [13-15] 和transcription start site (TSS)-associated RNAs (TSSaRNAs)。另外,还有来源于着丝粒异染色质区域的RNA[16]。
虽然导入外源siRNA 对靶基因进行沉默是分子生物学研究的重要方法,近年研究发现哺乳动物细胞中也存在内源的siRNA (endo-siRNAs),这些RNA 主要来源于细胞中转座元件、异染色质重复序列,或者是与mRNA 反义的假基因[17-18, 21]。microRNA和siRNA 在细胞质中通过组建RISC 复合物引导Argonaute 蛋白与特定区域的靶标RNA 结合实现对基因的沉默,但是Argonaute 蛋白在细胞核内的存在以及RNAi 通路在表观遗传调控上的作用,赋予了这套系统更复杂、多层面的功能。例如,在细胞周期基因POLR3D 启动子区域反义方向转录出的miR-320,顺式调控POLR3D基因表达,抑制其转录。miR-320 介导Argonaute-1 (AGO1) 蛋白与RRC2 复合物组分EZH2 的相互作用,导致POLR3D 基因启动子区域组蛋白H3 上第27 位赖氨酸的三甲基化(H3K27me3),从而抑制转录[19]。尽管大部分Argonaute 蛋白的表达和功能都与microRNA 以及siRNA 相关,但是还有另一类Argonaute 蛋白,称为PIWI 蛋白,参与生殖细胞的发育。PIWI 和PIWI相关蛋白与另一类特殊的小(26~30 nt) RNA 分子( 即piRNA) 相互作用,调节生殖细胞中转座子活性及染色质状态。假基因来源的endo-siRNA 在哺乳动物卵子中的功能类似于精子中的piRNA[20-21]。
2 长链非编码RNA对基因转录的调控
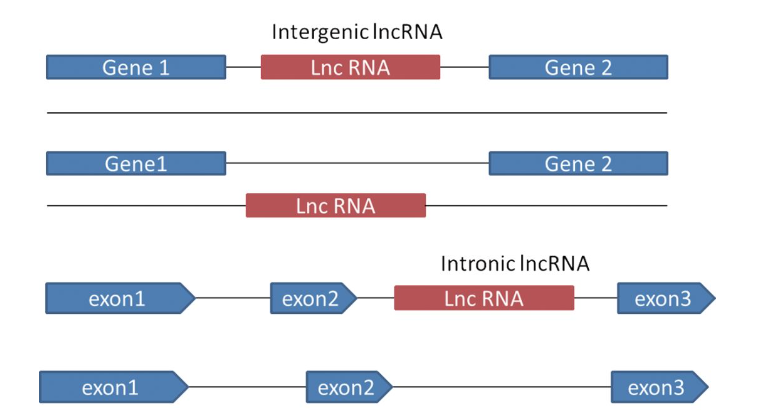
2005 年以来,像哺乳动物cDNA 功能注释等大规模cDNA 测序计划的进行,揭示了长链非编码RNA 在哺乳动物细胞中高丰度、远高于mRNA 种类的表达。长链非编码RNA 是长度大于200 nt,不具有编译蛋白质能力的一类RNA[20]。长链非编码RNA 通常根据它们在基因组上对应位置附近的蛋白质编码基因来分类。根据这一规则,可以将长链非编码RNA 分为4 大类,包括基因间长链非编码RNA (intergenic lncRNA,lncRNA 由基因组上编码基因之间的间隔序列转录产生,也即long intergenic ncRNA,lincRNA)、内含子长链非编码RNA (intronic lncRNA,即lncRNA 完全由蛋白质编码基因内的内含子转录而来)、正义长链非编码RNA (sense lncRNA,lncRNA 基因转录方向与蛋白质编码基因转录方向相同,并且与蛋白质编码基因外显子有部分或者全部重叠)、反义长链非编码RNA (antisense lncRNA, lncRNA 基因转录方向与蛋白质编码基因转录方向相反)[7,20]( 图1)。
人类基因组中大约有98%~99% 的非编码区域散布在基因组编码区域之间,小鼠基因组中87%的编码RNA 和与其反义的转录本同时存在,人类基因组中32% 存在这种情况[22]。
综述长链非编码RNA 的功能以及作用机制是一项很困难的任务。因为新的关于lncRNA 的文章层出不穷,lncRNA 的种类及功能时时在更新。另外,尽管我们习惯对每种lncRNA 的功能进行特定的分类,但是不能忽略的一点是,一条1 nt 左右的lncRNA 已足够长到可能具有多种功能,而有的时候在不同的组织中或者在发育的不同阶段有着不同的调控功能。lncRNA 可以直接在其转录位置附近起作用( 顺式作用, in cis),也可以对距离其转录位置很远的基因起调控作用( 反式作用,in trans)。目前研究发现的lncRNA 对转录调控的机制可以大致分为以下几类[7-11,22],见图2。下面将对这几类不同的调控机制分别详细介绍。
2.1 lncRNA直接调控转录因子复合物的装配或活性
IncRNA 可以通过调节转录激活因子、共调节因子的活性,也可以作为支架(scaffold) 募集转录调控因子,或者直接作用于RNA 聚合酶II 对基因的转录进行调控[7-11,22]。Alu RNA (280 nt) 是RNA聚合酶Ⅲ的转录产物,由在哺乳动物基因组中广泛存在的SINEs ( 在人中为Alu 序列,小鼠中为SINE序列) 元件转录而来。当细胞受到一系列的刺激( 如热激、病毒感染等) 或者发育信号调控时,AluRNA 在细胞中的表达量上调,并直接与RNA 聚合酶II 结合,妨碍RNA 聚合酶II 与DNA 模板的有效结合,抑制转录的进行[23]。7SK RNA 是另一个调控转录的lncRNA 的经典代表,其也是RNA 聚合酶Ⅲ的转录产物,长度331 nt,调控转录因子P-TEFb 的活性和作用效果。P-TEFb 是细胞周期依赖性蛋白激酶,可以磷酸化RNA 聚合酶II CTD(carboxy-terminal domain) 区域第2 位的丝氨酸,也可磷酸化激活转录延伸因子DSIF 以及NELF,这些磷酸化对转录延伸至关重要[24]。因为7SK RNA是在小核糖核蛋白复合体(snRNP) 中发现的,所以,归类为小核RNA (snRNA),但是由于其长度大于200 nt,所以作为lncRNA 介绍。7SK RNA 作为支架(scaffold),先与HEXIM1/2、LARP7 蛋白结合,形成抑制复合物。P-TEFb 与7SK 结合时,处于无活性状态,从而抑制了转录的延伸过程。最新的研究发现,一类DEAD-box helicase —DDX21 与7SKRNA 结合,将P-TEFb 从与7SK 的结合中释放出来,使其磷酸化RNA 聚合酶Ⅱ CTD 区域第2 位的丝氨酸[25]。lincRNA-p21 是一个基因间长链非编码RNA,长度约3.1 kb,p53 激活其转录,在DNA 损伤后作为p53 依赖的转录抑制的关键因子。p53 作
为细胞内重要的抑癌基因参与基因组稳定性的维持。DNA 发生损伤后,p53 蛋白会激活转录应答,激活或抑制相关基因转录。由p53 激活转录的基因中,包括大量的lincRNA,lincRNA-p21 就是其中一个。它与一个特殊的hnRNP 蛋白hnRNP-K 结合,hnRNP-K 在受p53 抑制的基因上有明显的定位区域, 所以, 目前认为p53 抑制基因表达是通过lincRNA-p21 与hnRNP-K 结合,hnRNP-K 与DNA上特定区域结合, 抑制转录。新的研究发现,linc-p21 与hnRNP-K 结合,在p53 依赖的基因表达调控途径中作为共刺激因子,促进邻近基因p21 的转录[26]。
2.2 通过对染色质上DNA或组蛋白进行表观遗传
修饰或直接改变染色体结构调控转录染色质的结构( 组成真核生物基因组的组蛋白和DNA 的组合形式) 影响DNA 与RNA 聚合酶Ⅱ以及转录因子的结合,对基因转录起着间接调控的作用。而染色体的结构以及活性会受到组蛋白或者DNA 的甲基化或乙酰化水平的影响,如基因启动子区H3K4me3 以及H3K36me3 与基因转录激活相关,H3K27me3 和H4K20me3 与转录抑制相关[27-28]。一些lncRNA 主要是以影响DNA 或组蛋白表观遗传修饰或改变染色体结构来调控基因转录的。
2.2.1 DNA 甲基化
DNA 的甲基化是第一种被发现的表观遗传调控机制。典型的DNA 甲基化发生在基因组CpG 上,而位于转录起始位点附近的CpG 群却是非甲基化的,这个区域就是CpG 岛。以往研究发现的会影响DNA 甲基化的RNA 大都为编码蛋白质的mRNA的反义转录产物,如HBA1 (haemoglobin α1) 的反义
RNA,LUC7L 和p53 的反义RNA,wrap53[29]。LUC7L通过甲基化启动子区CpG 岛抑制基因转录,而wrap53 则是与CTCF 结合,调节p53 基因的表达。已知的与DNA 甲基化有关的lncRNA 并不多,其中还有Arin 和ecCEBPA。118 kb 长的“巨型”Arin (antisense to insulin-like growth factor 2 receptor)(Igf2r) 的转录与Igf2r 基因的启动子区有重叠,干扰了RNA 聚合酶Ⅱ在Igf2r 启动子区的结合,导致Igf2r 基因的沉默。Arin 还可以通过招募H3K9 甲基转移酶沉默Slc22a3 基因的表达[30]。ecCEBPA (extra-coding CEBPA) 在距离CEBPA 基因转录起始位点上游大约2 kb 的位置起始转录,转录方向与CEBPA 基因转录方向相同,长度约为4.5 kb [31-32]。
2.2.2 组蛋白修饰
参与组蛋白修饰的酶是作为复合物起作用的,识别( 阅读器,reader)、添加( 书写器,writer)、去除( 擦除器,erasers) 或替换各种类型的修饰。典型的起书写作用的酶,包括EZH2 和HMT 等。EZH2是PRC2 ( polycomb repressive complex 2) 复合物的催化亚基,与H3K27me3 有关,HMT 作为甲基转移酶,催化H3K9me2/3 的产生。擦除器的代表是LSD1 去甲基化酶,能特异去除某些组蛋白修饰标记。阅读器、书写器以及擦除器三者紧密合作[33-34]。组蛋白修饰酶是如何特异识别并结合在它们的目标位点上的并没有清楚的定论,有假设认为各种非编码RNA 在靶基因位点特异募集组蛋白修饰酶的过程中起重要作用[35]。
长链非编码RNA 调控基因转录的一个重要的方式,是作为支架(scaffold) 招募组蛋白修饰相关的复合物,对染色质组蛋白进行甲基化或乙酰化修饰,抑制或激活基因的转录。最经典的例子是与哺乳动物X 染色体随机失活相关的XIST,它是第一个被发现的lncRNA ( 也有认为H19 是第一个被发现的lncRNA,但是随后的研究表明,H19 很可能是一个microRNA 的大前体)。雌性细胞中两条染色体其中一条表达Xist RNA。Xist RNA 导致表达它的那条X 染色体失活的机制是,招募PRC2 复合物组蛋白甲基转移酶,移除H3K27me3 标志,抑制转录[36]。很多lncRNA,特别是在细胞中只有一个或者几个拷贝的那些lncRNA,作用方式是顺式的,即这些lncRNA 是在它们转录位点附近起作用的。也有一些lncRNA 作用于转录位点以外的基因,即反式作用,比如HOTAIR,这些lncRNA 作用的机制可能是通过lncRNA-mRNA 互补配对,lncRNADNA形成三聚体[37],或者与后来提出的启动子相关的pRNA 募集DNMT3b 沉默rRNA 基因的机制类似[38]。通过招募组蛋白修饰酶影响基因转录的lncRNA 有很多,如HOTAIR (Hox anti-sense intergenic RNA)、HOTTIP (HOXA transcript at the distal tip)、BDNF-AS[41] 等。HOTAIR,大约2.2 kb,由人HOXC位点转录而来,5' 端结合PRC2 复合物的SUZ12 亚基,而3' 端可以结合LSD1/CoREST/REST 复合物,将这些组蛋白修饰酶招募到HOXD 位点,导致H3K27me3 产生和H3K4 的去甲基化,抑制转录[39]。与HOTAIR 相反,HOTTIP 通过调节染色质结构激活基因转录。HOTTOP 是由哺乳动物HOXA 基因一端转录出的,长约3.7 kb 的非编码RNA,结合MLL1/WDR5 复合物,MLL1/WDR5 复合物作为书写器,修饰组蛋白,产生H3K4me3 和H3K4me2甲基化,激活HOXA 基因转录[40]。
2.3 enhancer RNA
转录增强子元件转录而来的ncRNA ( 称为eRNA) 是近年新发现的。以往认为,每个增强子DNA 元件可以结合特异的蛋白质,这些蛋白质通过蛋白质直接相互作用,与启动子元件相互作用,形成一个大的DNA 环状结构,距离启动子很远的增强子元件通过这样的方式促进转录的进行。eRNA 的发现导致了一个新的模型的形成,即这类增强子转录出的ncRNA 与中介复合物(mediator complex) 结合,促进转录起始[42,9]。目前关于eRNA在转录调控中的功能研究还刚刚开始。
2.4 作为诱饵分子(Decoy)
lncRNA 的这类功能与上述所述的功能不同,不是募集或组织蛋白质复合物,而是作为诱饵,与蛋白质结合,抑制蛋白质的功能。小鼠中发现的B2 RNA 以及在人类细胞中存在的Alu、Gas5、7SK等RNA 都可以属于这类分子。B2 RNA、Alu RNA受一系列细胞应激反应或发育信号的调节,它们定位在目标基因的启动子区,可以直接与RNA 聚合酶II 结合,抑制聚合酶的磷酸化激活,从而抑制转录的进行[43-44]。Gas5 RNA 直接与肾上腺皮质激素受体(GR) 的DNA 结合结构域结合,与Gas5结合的GR 无法与基因组上GR 应答元件结合激活转录,从而抑制了肾上腺皮质激素依赖的转录过程的进行[45]。
3 环状非编码RNA对基因转录的调控
环状RNA (circ RNA) 很早就被发现存在于细胞中,最开始被发现的环状RNA 中,被大家熟知的一个circRNA 是circSRY[46]。尽管很早就被研究人员发现,但是它们的功能一直是未知的。近年来,人们开始关注这类特殊的ncRNA,研究结果发现这类ncRNA 具有很重要的功能( 图3)。两个circRNA被发现是作为microRNA“海绵”(sponge),其中CDRIas 上有63 个miR-7 结合位点,结合miR-7,从而封闭了miR-7 的作用;而circSRY 可以作为miR-138 的海绵 [47-48]。还有一些circRNA 具有调控转录的功能:一类内含子来源的环状RNA (ciRNA)可能与延伸状态的RNA 聚合酶II 结合,以未知的机理顺式调控其基因的转录[49] ;一类外显子- 内含子circRNAs (EIciRNAs),主要定位在细胞核中,与U1 snRNP 发生相互作用促进了它们亲本基因(parental gene)的转录;一类只含有内含子的环状RNA (circular intronic RNA),可以顺式调控RNA 聚合酶II 转录的延伸[50]。对于环状RNA 功能的研究仍然是一项重要的任务,随着研究的深入,相信这类特殊的非编码RNA 的神秘面纱将被逐渐揭开。
4 展望
非编码RNA 作为哺乳动物基因转录的关键调控因子,逐渐被大家认可。尽管有很多非编码RNA的功能被发现并证实,但是这仅仅是一小部分,还有很多非编码RNA 的功能是未知的,或者已知功能的非编码RNA 还可能会存在其他的功能。随着研究的进行,科研技术的发展,还会有更多的新类型的非编码RNA 被发现。另外一个问题是,通过实验及高通量测序技术测得的一系列与蛋白质结合的非编码RNA 数据只能说明相互作用的存在,但是究竟是如何相互作用,相互作用产生怎样的结果,仍然需要更多的研究。非编码RNA 从最开始被认为是无用的转录副产物,到现在被发现具有如此多的重要调控功能,足以证明非编码RNA 是一个巨大的宝库,对非编码RNA 的研究时刻都会有惊喜出现。
[参 考 文 献]
[1] Mattick JS, Makunin IV. Non-coding RNA. Hum Mol
Genet, 2006, 15: 17-29
[2] Mattick JS. Non-coding RNAs: the architects of
eukaryotic complexity. EMBO Rep, 2001, 2: 986-91
[3] Carninci P. The transcriptional landscape of the
mammalian genome. Science, 2005, 309: 1559-63
[4] Ponjavic J, Ponting CP, Lunter G. Functionality or
transcriptional noise? Evidence for selection within long
noncoding RNAs. Genome Res, 2007, 17: 556-65
[5] Ponting CP, Oliver PL, Reik W. Evolution and functions
of long noncoding RNAs. Cell, 2009, 136: 629-41
[6] Eddy SE. Non–coding RNA genes and the modern RNA
world. Nat Rev Genet, 2001, 2: 919-29
[7] Ma L, Bajic VB, Zhang Z. On the classification of long
non-coding RNAs. RNA Biol, 2013, 10: 925-33
[8] Wilusz JE, Sunwoo H, Spector DL. Long noncoding
RNAs: functional surprises from the RNA world. Genes
Dev, 2009, 23: 1494-504
[9] Cech TR, Steitz1 JA. The noncoding RNA revolution—
trashing old rules to forge new ones. Cell, 2014, 157: 77-
94
[10] Goodrich JA, Kugel JF. Non-coding-RNA regulators of
RNA polymerase II transcription. Nat Rev Mol Cell Biol,
2006, 7: 612-6
[11] Kugel JF, Goodrich JA. Non-coding RNAs: key regulators
of mammalian transcription. Trends Biochem Sci, 2012,
37: 144-51
[12] Taft RJ, Simons C, Nahkuri S, et al. Nuclear-localized tiny
RNAs are associated with transcription initiation and
splice sites in metazoans. Nat Struct Mol Biol, 2010, 17:
1030-4
[13] Fejes-Toth K, Sotirova V, Sachidanandam R, et al. Posttranscriptional
processing generates a diversity of
5′-modified long and short RNAs. Nature, 2009, 457:
1028-32
[14] Han J, Kim D, Morris KV. Promoter-associated RNA is
required for RNA-directed transcriptional gene silencing
in human cells. Proc Natl Acad Sci USA, 2007, 104:
12422-7
[15] Kapranov P, Willingham AT, Gingeras TR. Genome-wide
transcription and the implications for genomic
organization. Nat Rev Genet, 2007, 8: 413-23
[16] Reinhart BJ, Bartel DP. Small RNAs correspond to
centromere heterochromatic repeats. Science, 2002, 297:
1831-31
[17] Nilsen TW. Endo-siRNAs: yet another layer of complexity
in RNA silencing. Nat Struct Mol Biol, 2008, 15: 546-8
[18] Okamura K, Lai EC. Endogenous small interfering RNAs
in animals. Nat Rev Mol Cell Biol, 2008, 9: 673-78
[19] Kim DH, Saetrom P, Snøve O, et al. MicroRNA-directed
transcriptional gene silencing in mammalian cells. Proc
Natl Acad Sci USA, 2008, 105: 16230-5
[20] Luteijn MJ, Ketting RF. PIWI-interacting RNAs: from
generation to transgenerational epigenetics. Nat Rev
Genet, 2013, 14: 523-34
[21] Kim VN, Han J, Siomi MC. Biogenesis of small RNAs in
animals. Nat Rev Mol Cell Biol, 2009, 10: 126-39
[22] Rinn JL, Chang HY. Genome regulation by long
noncoding RNAs. Annu Rev Biochem, 2012, 81: 145-66
[23] Mariner PD, Walters RD, Espinoza CA, et al. Human Alu
RNA is a modular transacting repressor of mRNA
transcription during heat shock. Mol Cell, 2008, 29: 499-
509
[24] Nguyen VT, Kiss T, Michels AA, et al. 7SK small nuclear
RNA binds to and inhibits the activity of CDK9/cyclin T
complexes. Nature, 2001, 414: 322-5
[25] Diribarne G, Bensaude O. 7SK RNA, a non-coding RNA
regulating P-TEFb, a general transcription factor. RNA
Biol, 2009, 6: 122-8
[26] Dimitrova N, Zamudio JR, Jong RM, et al. LincRNA-p21
activates p21 in cis to promote Polycomb target gene
expression and to enforce the G1/S checkpoint. Mol Cell, 2014, 54: 777-90
[27] Berger SL. Histone modifications in transcriptional
regulation. Curr Opin Genet Dev, 2002, 12: 142-8
[28] Zilberman D, Gehring M, Tran RK, et al. Genome-wide
analysis of Arabidopsis thaliana DNA methylation
uncovers an interdependence between methylation and
transcription. Nat Genet, 2007, 39: 61-9
[29] Saldaña-Meyer R, González-Buendía E, Guerrero G, et al.
CTCF regulates the human p53 gene through direct
interaction with its natural antisense transcript, Wrap53.
Genes Dev, 2014, 28: 723-34
[30] Latos PA, Pauler FM, Koerner MV, et al. Airn
transcriptional overlap, but not its lncRNA products,
induces imprinted Igf2r silencing. Science, 2012, 338:
1469-72
[31] Di Ruscio A, Ebralidze AK, Benoukraf T, et al. DNMT1-
interacting RNAs block gene-specific DNA methylation.
Nature, 2013, 503: 371-6
[32] Nagano T, Mitchell JA, Sanz LA, et al. The Air noncoding
RNA epigenetically silences transcription by targeting
G9a to chromatin. Science, 2008, 322: 1717-20
[33] Falkenberg KJ, Johnstone RW. Histone deacetylases and
their inhibitors in cancer, neurological diseases and
immune disorders. Nat Rev Drug Discov, 2014, 13: 673-
91
[34] Chi P, Allis CD, Wang GG. Covalent histone modifications:
miswritten, misinterpreted, and miserased in human
cancers. Nat Rev Cancer, 2010, 10: 457-69
[35] Tsai MC, Manor O, Wan Y. Long noncoding RNA as
modular scaffold of histone modification complexes.
Science, 2010, 329: 689-93
[36] Zhao J, Sun BK, Erwin JA, et al. Polycomb proteins
targeted by a short repeat RNA to the mouse X chromosome.
Science, 2008, 322: 750-6
[37] Vance KW, Ponting CP. Transcriptional regulatory
functions of nuclear long noncoding RNAs. Trends Genet,
2014, 30: 348-55
[38] Schmitz KM, Mayer C, Postepska A, et al. Interaction of
noncoding RNA with the rDNA promoter mediates
recruitment of DNMT3b and silencing of rRNA genes.
Genes Dev, 2010, 24: 2264-9
[39] Tsai MC, Manor O, Wan Y, et al. Long noncoding RNA as
modular scaffold of histone modification complexes.
Science, 2010, 329: 689-93
[40] Wang KC, Yang YW, Liu B, et al. A long noncoding RNA
maintains active chromatin to coordinate homeotic gene
expression. Nature, 2011, 472: 120-4
[41] Modarresi F, Faghihi MA, Lopez-Toledano MA, et al.
Inhibition of natural antisense transcripts in vivo results in
gene-specific transcriptional upregulation. Nat Biotechnol,
2012, 30: 453-9
[42] Kim TK, Hemberg M, Gray JM, et al. Widespread
transcription at neuronal activity-regulated enhancers.
Nature, 2010, 465: 182-7
[43] Mariner PD, Walters RD, Espinoza CA, et al. Human Alu
RNA is a modular transacting repressor of mRNA
transcription during heat shock. Mol Cell, 2008, 29: 499-
509
[44] Espinoza CA, Allen TA, Hieb AR, et al. B2 RNA binds
directly to RNA polymerase II to repress transcript
synthesis. Nat Struct Mol Biol, 2004, 11: 822-9
[45] Kino T, Hurt DE, Ichijo T, et al. Noncoding RNA Gas5 is
a growth arrest and starvation-associated repressor of the
glucocorticoid receptor. Sci Signal, 2010, 3: ra8
[46] Dubin R A, Kazmi MA, Ostrer H. Inverted repeats are
necessary for circularization of the mouse testis Sry
transcript. Gene, 1995, 167: 245-8
[47] Hansen TB, Jensen TI, Clausen BH, et al. Natural RNA
circles function as efficient microRNA sponges. Nature,
2013, 495: 384-8
[48] Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs
are a large class of animal RNAs with regulatory potency.
Nature, 2013, 495: 333-8
[49] Zhang Y, Zhang XO, Chen T, et al. Circular intronic long
noncoding RNAs. Mol Cell, 2013, 51: 792-806
[50] Li ZY, Huang C, Bao C, et al. Exon-intron circular RNAs
regulate transcription in the nucleus. Nat Struct Mol Biol,
2015, 22: 256-64

