随着AlphaFold在蛋白质结构预测精度上取得了革命性进步,RNA结构预测自然变成下一个急需解决的重大科学问题。最新的国际结构预测大会表明人工智能和深度学习 对RNA结构预测没有显著帮助,RNA结构预测准确度依然较低。这里面主要的原因有两点:
1. 相对与蛋白质有大量的三维结构,独立功能RNA的三维结构目前为止不足200个。
2. 相对于蛋白质的结构域和功能单元研究,RNA motif 的研究严重不足。
kink turn (k-turn)是一种广泛存在的RNA motif,可在双链RNA中产生紧密扭结,从而介导三级相互作用。它参与细胞中许多关键 RNA 结构的构建,这些结构对于翻译、RNA 加工和遗传控制都非常重要[1]。k‐junction是k‐turn的一种复杂结构形式,与k‐turn相比具有额外的第三个螺旋。之前本课题组研究表明,k-turn的折叠特性和构象取决于它们的局部序列,并阐明了一系列用于从序列预测这些特性的规则[2]。详细研究RNA motif,对RNA结构进行系统分析和比较,有助于提高我们从序列中预测RNA精确三维结构的能力。

k-turns的结构包括一个短的(通常是3nt)凸起环,后面是保守的G:A和A:G碱基对(图1A)。它可以通过诱导折叠(例如添加金属离子)形成一个紧密弯曲的结构,夹角约50°,由2′羟基和G:A碱基对中A的氮原子之间的关键交叉链氢键稳定[3]。k‐junctions作为k‐turns的一种复杂形式,保留串联的G:A碱基对和交叉链氢键,但在n(非凸起)链上具有额外的第三个螺旋。k‐junctions结构首次在拟南芥和大肠杆菌TPP核糖开关的结构中被发现(图1B和C)[4-5]。2017年Weinberg等人利用生物信息学鉴定出224个新的结构性RNA motif(structured noncoding RNA)[6]。通过对他们的二级结构进行分析,研究者确定了四个可能的k‐turn结构,并在溶液和晶体结构中进行了实验证明[7]。此外,研究者还在DUF-3268序列中发现了一个可能的k‐junction(图1D)。
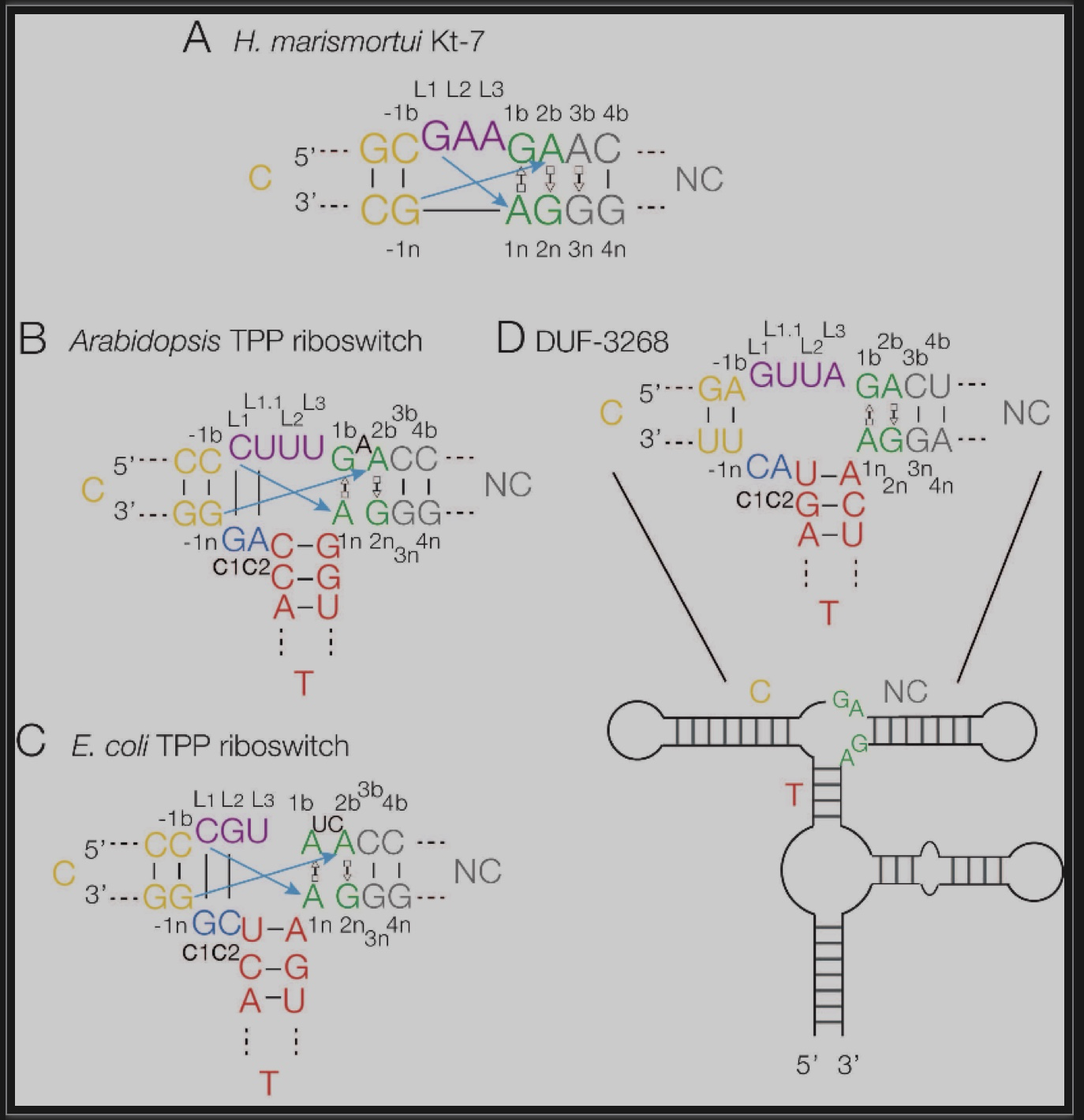
图1 核糖开关k‐junctions和DUF - 3268的序列
TPP核糖开关的k‐junctions和标准k‐turns之间的二级结构对比表明,很可能k‐junctions在添加金属离子后会折叠成和k‐turns类似的结构,然而这在实验中从未被证实。研究者通过荧光共振能量转移(FRET)研究预期的离子诱导的k‐junctions折叠(图2A)。在两个k‐junctions中添加Mg2+都会显著增加FRET效率(图2B和C);和k‐turns类似,两种k‐junctions也对一价金属离子也能响应,随着Na+的添加,FRET效率也随之增加(图2D和E),类似于标准k‐turns的折叠过程。
在标准的k‐turns中,L1的O2'提供的氢键对于离子诱导的折叠尤为重要,L1 O2'H突变体的折叠能力严重受损。研究者构建了拟南芥和大肠杆菌k‐junctions相应的L1 O2'H突变体,并进行Mg2+滴定实验(图2B和C)。两种k‐junctions变体在添加Mg2+后都无法很好地折叠。这证明了跨链氢键对于k‐junctions折叠的重要性。
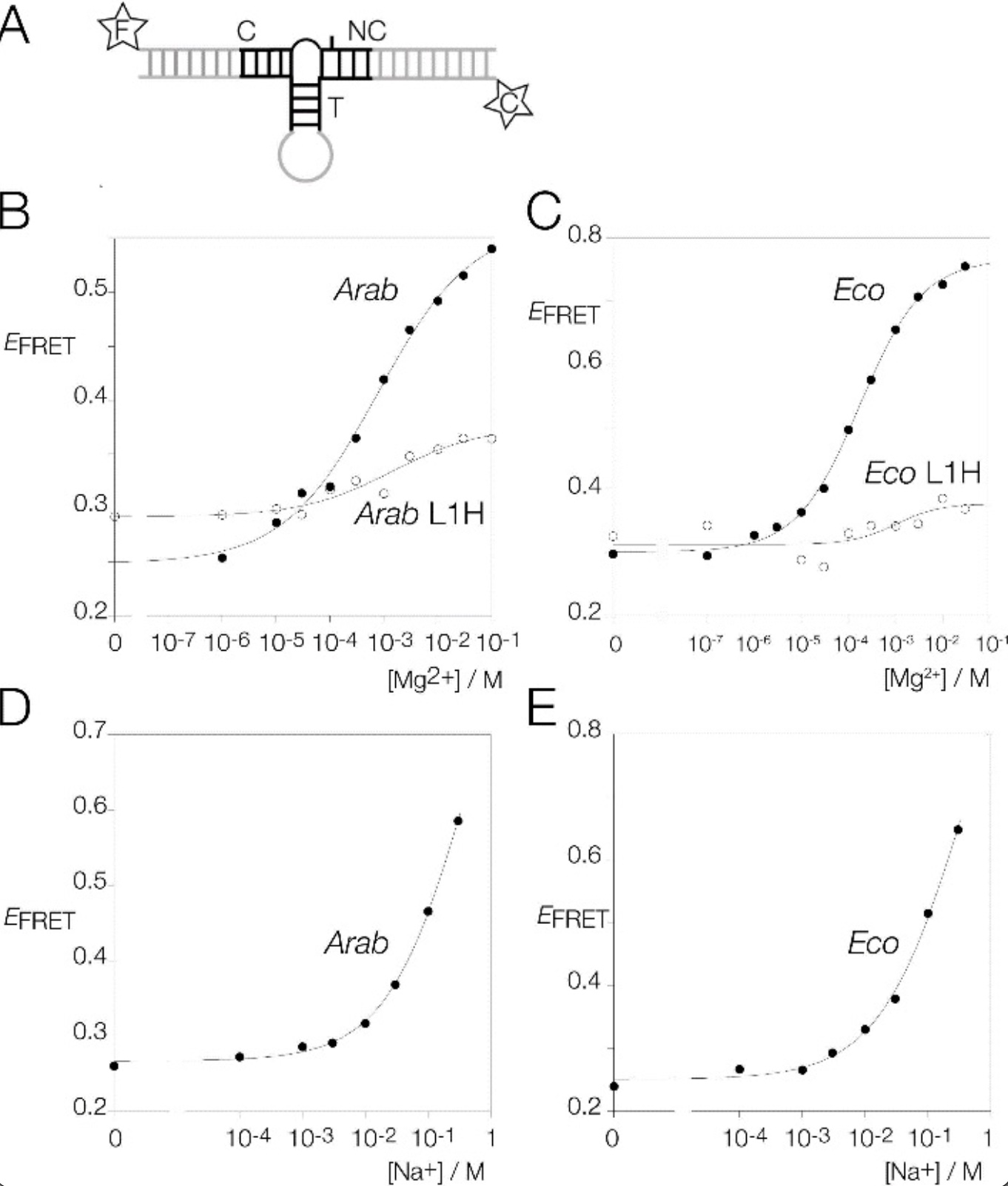
图2 金属离子诱导拟南芥和大肠杆菌TPP核糖开关k‐junctions折叠的FRET分析
接着研究者对DUF - 3268中可能的k‐junction进行离子诱导折叠。添加Mg2+或Na+到DUF-3268 k‐junction中会导致FRET效率的增加(图3A和B)。该k‐junction明显折叠成弯曲的几何结构,以响应金属离子的添加,但其特性与拟南芥和大肠杆菌的k‐junction明显不同。DUF-3268 k‐junction的EFRET起始值相比较高。同时也测试了DUF-3268 k‐junction可能的L1 O2'原子突变的影响,将Mg2+滴定到L1 O2'H突变体中导致初始EFRET值较低,并且折叠程度非常小(图3A)。
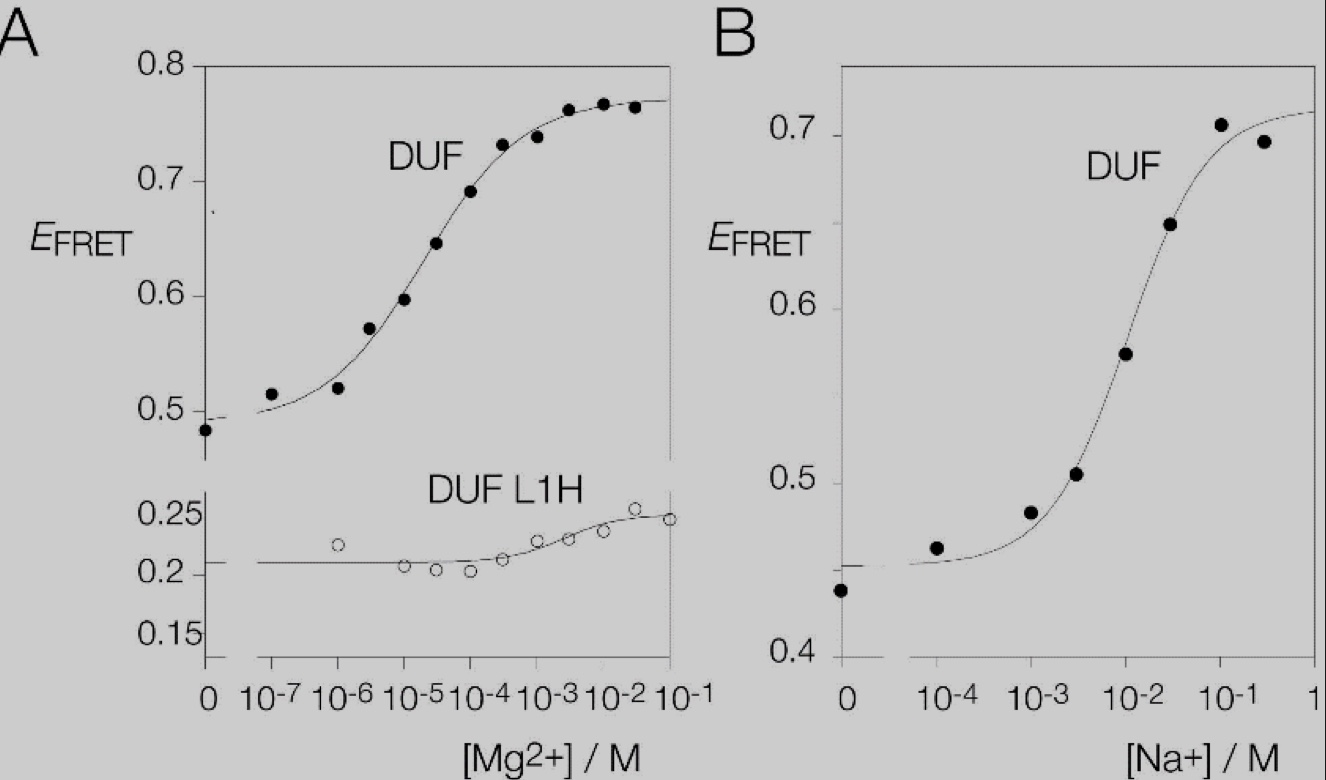
图3 金属离子诱导DUF‐3268 k‐junction折叠的FRET分析
研究者通过X-射线晶体学解析了DUF‐3268 k‐junction的晶体结构,分辨率为1.94 Å。DUF-3268 k‐junction的整体结构与拟南芥和大肠杆菌的k‐junction非常相似,形成一个三向螺旋连接。图4A展示了二级结构示意图,图4B和C展示了k‐junction的核心结构。两个非经典碱基对G1b:A1n和A2b:G2n均为trans G(sugar):A(Hoogsteen),与标准k‐turn一样。它们参与了标准的交叉链氢键(图4C、D和E)。如预测的被指定为L1的核糖的O2'给A1n N1提供了一个氢键(图4C和D)。类似地,被指定为-1n的核糖的O2'给A2b N1提供了一个氢键(图4C和E)。这与上面显示的原子突变体的折叠数据完全一致,晶体学结构证实了它们在k‐junction中的作用。RNA的构象表明其为N1型k‐turn,即A2b的N1位置接受一个质子。
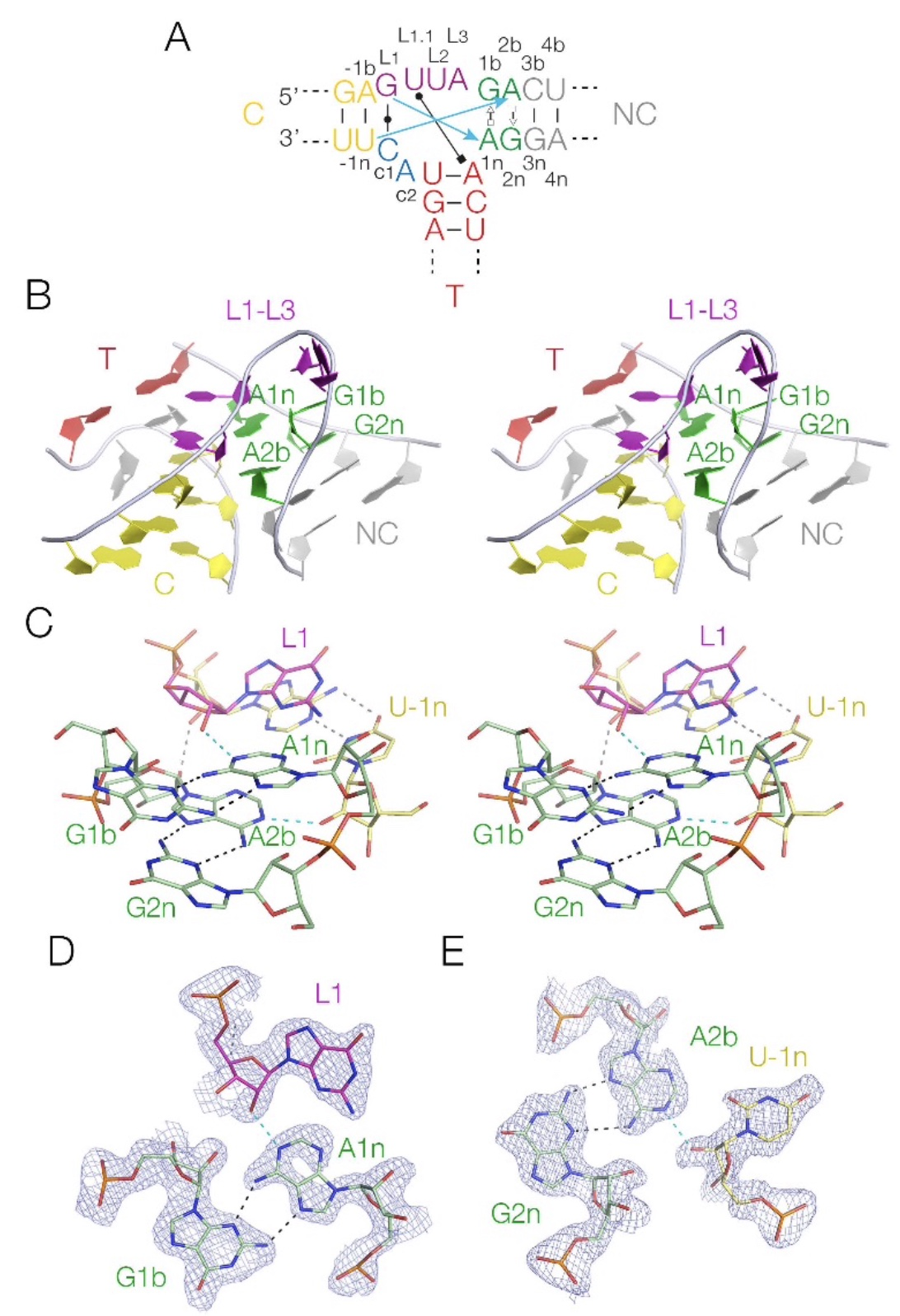
图4 DUF‐3268 k‐junction的晶体结构
DUF-3268 k‐junction与拟南芥和大肠杆菌的k‐junction之间最显著的差异在于G1b和A2b之间没有突出的核苷酸。因此研究者猜测拟南芥和DUF-3268的k‐junctions之间的折叠特性差异来源于前者突出的腺嘌呤。研究制备了一个去除A突出的拟南芥k‐junction突变体和一个在G1b和A2b之间插入A的DUF-3268 k‐junction突变体。通过测量随着Mg2+浓度变化而变化的EFRET来分析它们(图5)。从结果中可以看出在G1b和A2b之间存在突出核苷酸时,离子诱导的折叠受到影响;而当突出核苷酸不存在时,离子诱导的折叠显著改善。
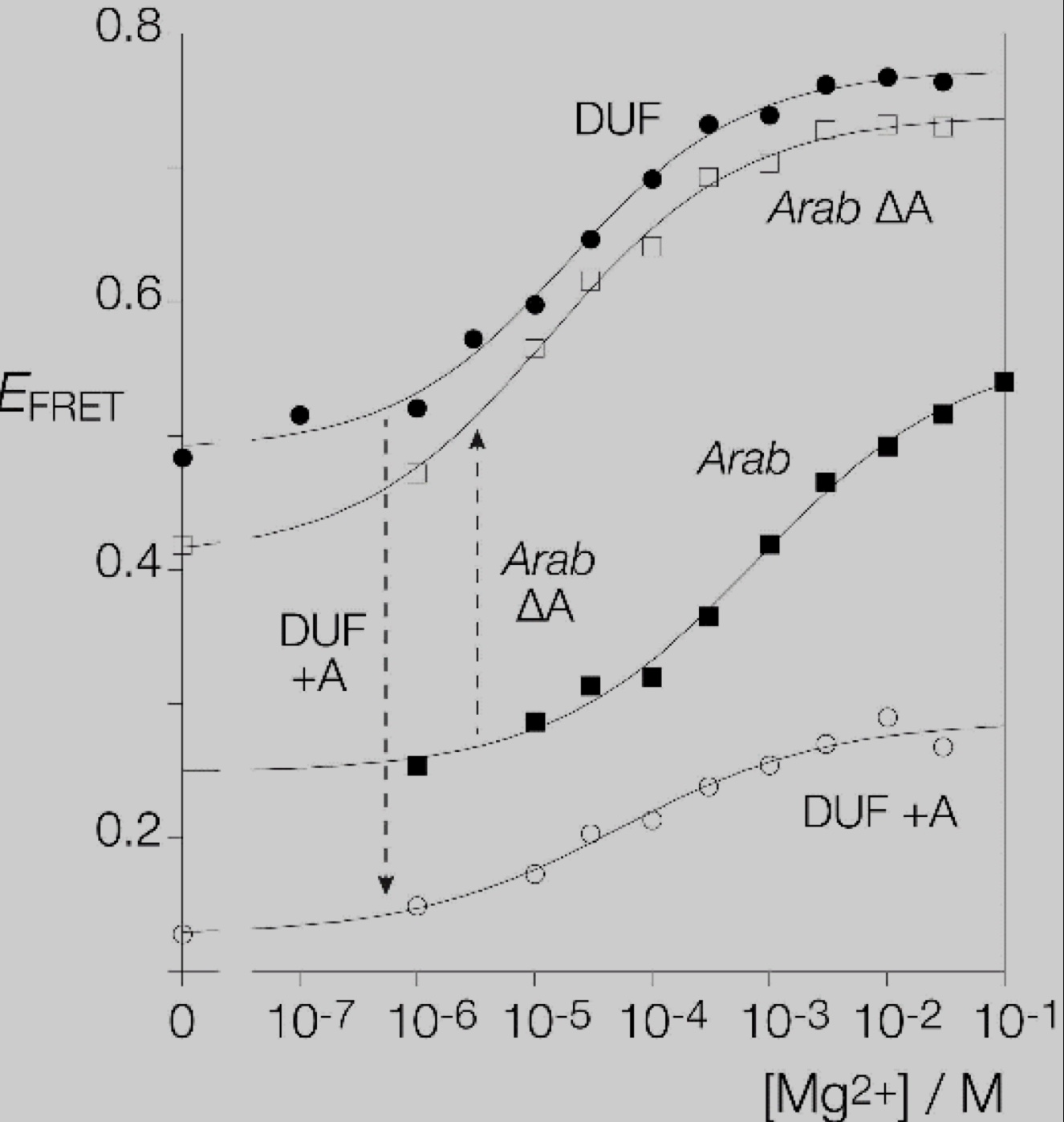
图5 插入在G1b和A2n之间的凸起腺嘌呤对拟南芥和DUF - 3268 k‐junction折叠的影响
由于DUF-3268采用了k‐junction构象,研究者想知道它是否在功能上可以替代大肠杆菌TPP核糖开关中的k‐junction。该研究设计了两个突变体:一是用DUF-3268 k‐junction替换了大肠杆菌TPP核糖开关的自然k‐junction序列;二是在DUF k‐junction序列中进行了A1nU点突变,已知该位点的突变会影响折叠(图6A)[8]。然后通过聚丙烯酰胺凝胶电泳和等温滴定量热法去检测添加TPP后是否会导致嵌合体核酸开关构象的改变。凝胶电泳和ITC的结果表明,DUF-3268可以在TPP核糖开关中功能替代大肠杆菌k‐junction结构,但其与配体的结合能力较原始序列弱(图6B-E)。这表明,替代的结构之间的微小构象差异对配体结合和核糖开关的整体构象具有显著影响。
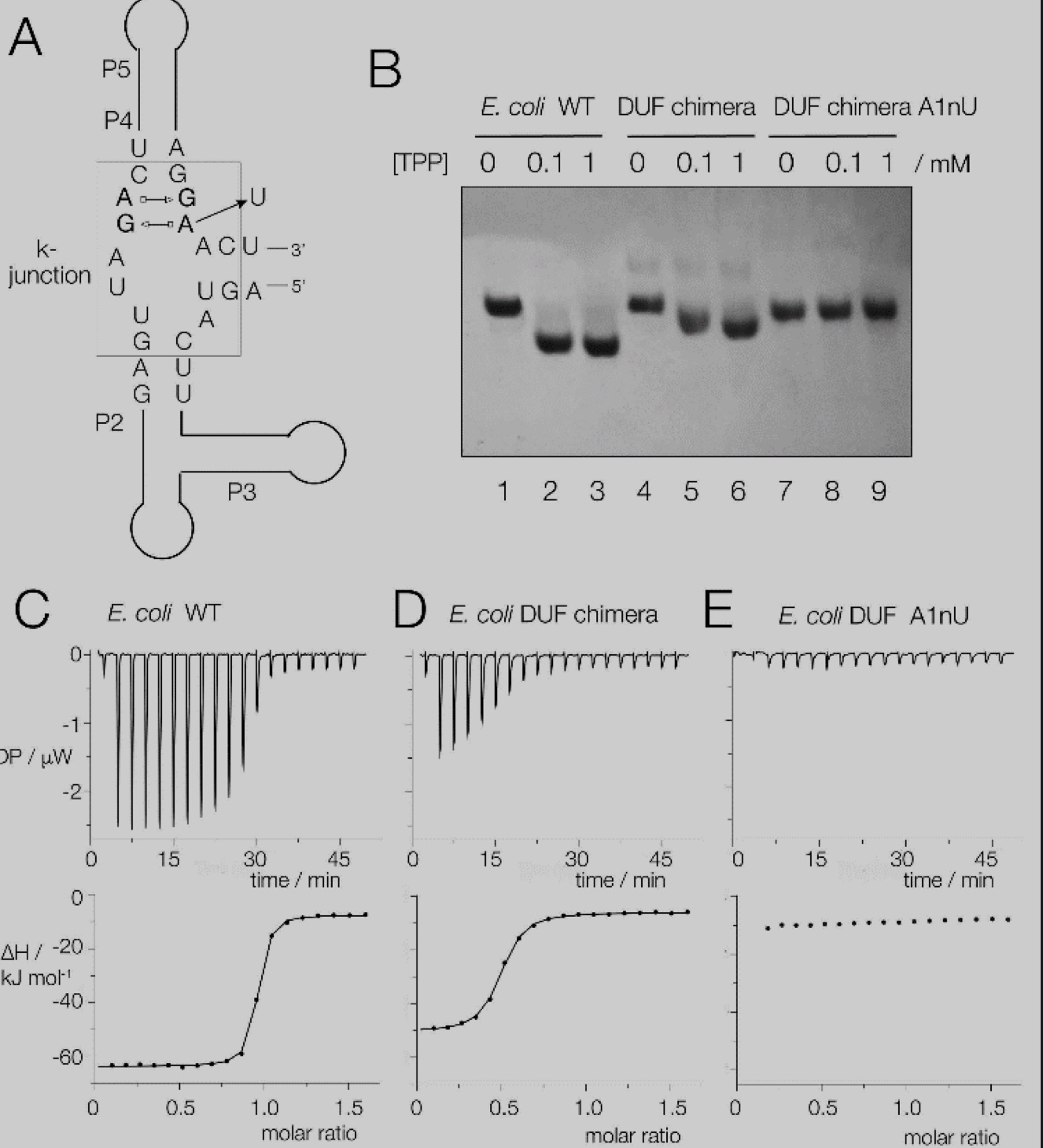
图6 在大肠杆菌TPP核糖开关中,天然k‐junction可以被DUF - 3268 k‐junction序列在功能上取代
研究这些构象特性以及它们如何受到局部序列突变的影响是非常重要的。这些信息对于从序列预测RNA的三维结构至关重要。此外,在RNA设计方面,随着对RNA motif的理解越来越深入,k-turn、k-junction和其他RNA motif的组合无疑将产生令人激动和新颖的RNA结构。

