作为适应性免疫系统中的核心成员,抗体在对付病原体方面发挥着至关重要的作用。在抗体产生过程中,初级抗体库是通过V(D)J重排产生的,对抗原的亲和力相对较低。之后,由AID胞苷脱氨酶启动的体细胞超突变(SHM)会在抗体编码序列中引入突变,提升抗体对抗原的亲和力,从而实现抗体的亲和力成熟。
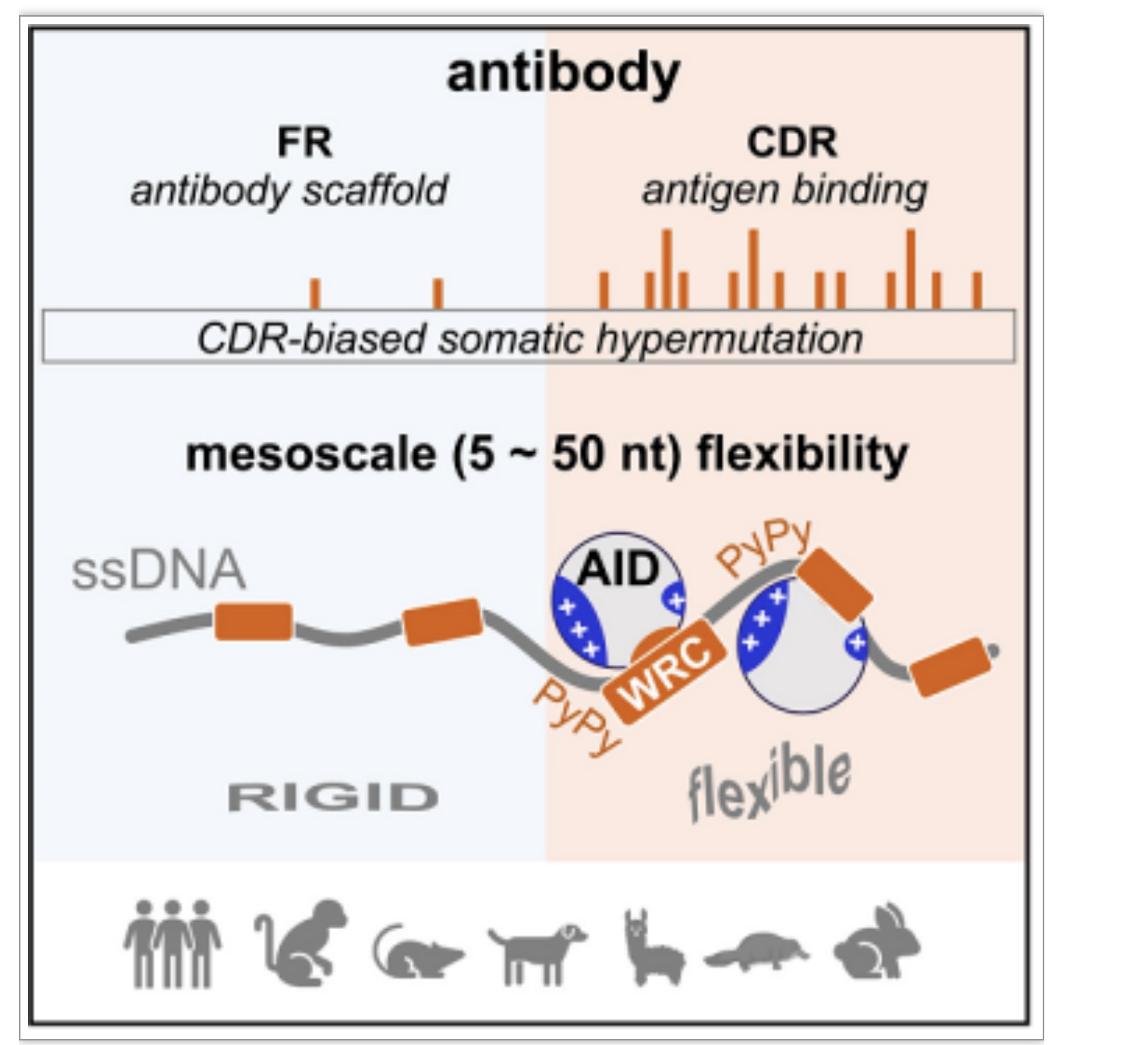
之前的研究发现,超突变在抗体可变区的外显子上并非均匀分布,而是集中在三个非连续的互补决定区(CDR)。早在20世纪80年代,科学家们便提出了超突变为什么具有偏好性的问题,但四十多年来,这仍然是一个谜。
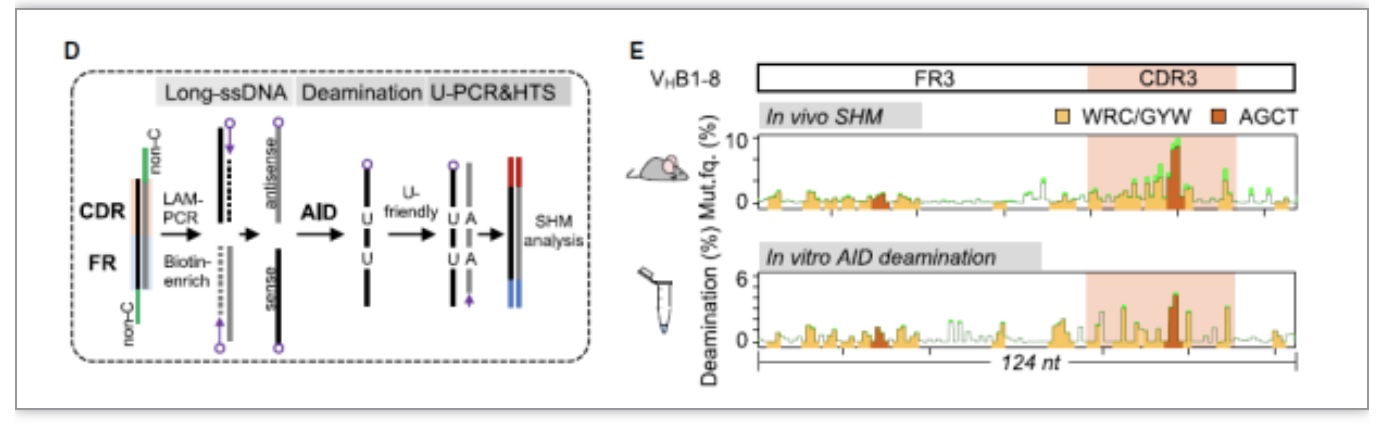
研究人员从经典的生化方法出发,联合高通量测序技术,建立了体外检测AID脱氨酶活性的新方法(图1)。他们使用了一段124-nt的单链DNA底物,覆盖小鼠VHB1-8外显子中的FR3-CDR3序列,这段序列在体内表现出WRC突变偏好。他们发现,体内CDR高突变特征可以在体外AID脱氨酶活分析中完全重现。
图1 体外AID脱氨酶活分析重现了体细胞超突变图谱
接下来,他们将生化分析的DNA底物扩展到27种有颌脊椎动物的1084条抗体基因序列。他们发现,在使用体细胞超突变作为抗体多样化策略的动物中,包括人、恒河猴、食蟹猴、小鼠、大鼠、狗、鸭嘴兽及羊驼等,CDR突变偏好高度保守。相比之下,一些采用基因转换机制的物种,如鸡、牛、羊和猪等,则并未表现出如此强烈的偏好。
周围序列背景决定了WRC基序的突变性
那么,这种突变偏好是否受DNA序列背景的影响?为了检验这一点,研究人员构建了一个携带IgVH-乱序等位基因的小鼠模型,其中每个CDR或FR的一个WRC(AGCT/AGCA)基序被保留在相同位置,而六个WRC基序周围的序列被随机置换。他们发现,在序列改变后突变频率发生了显著变化。这表明不同的DNA序列背景中可能存在中尺度的元件,增强或抑制WRC突变。
于是,他们利用CRISPR-Cas9技术构建了11种携带不同CDR3序列背景的小鼠模型。他们发现,其中6个模型的C/G核苷酸突变频率降低,而1个模型的突变频率升高。值得注意的是,序列改变越靠近WRC基序,则对该位点的突变频率影响越大。这些结果表明,周围序列背景决定了体内WRC基序的突变程度。
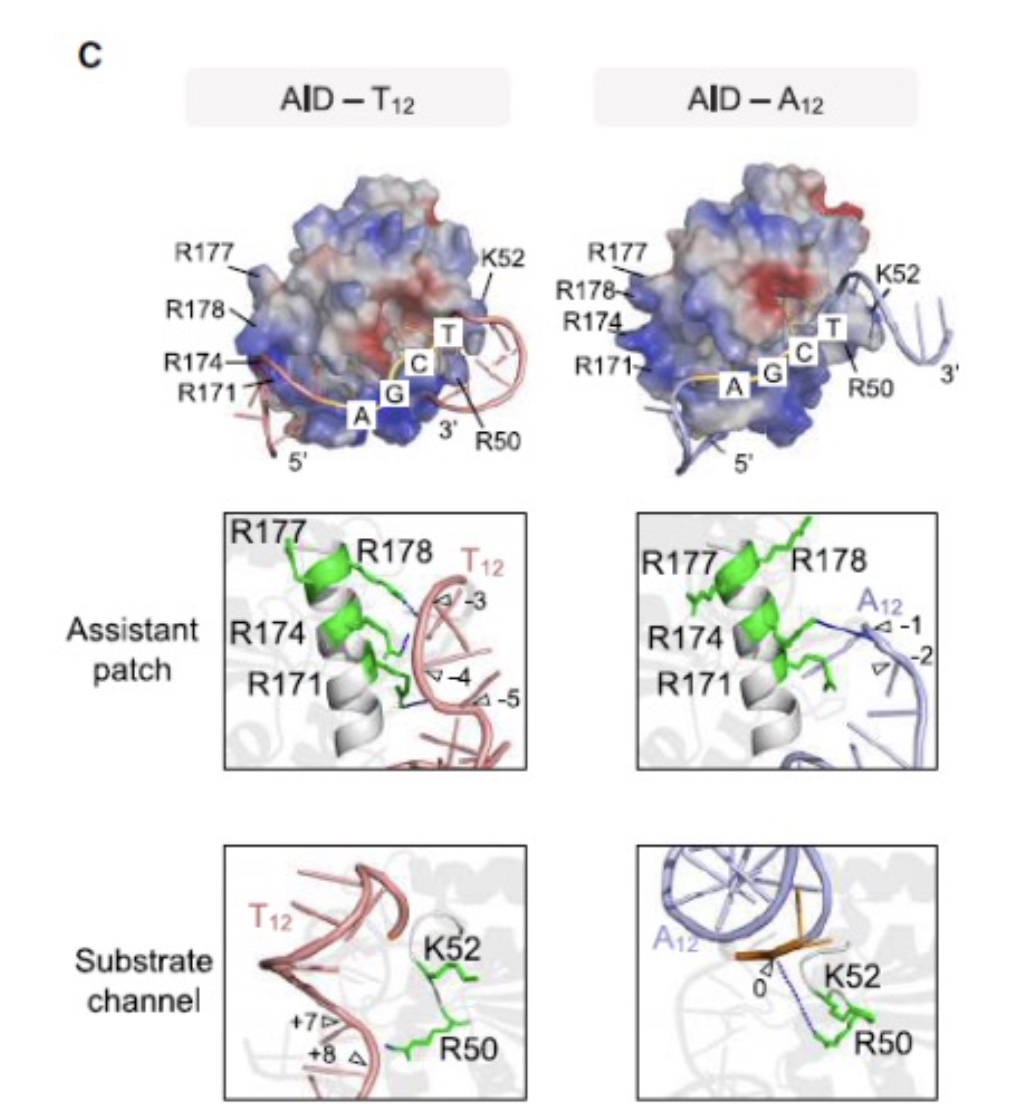
为了绘制AID和单链DNA底物的潜在相互作用,研究人员在poly(dT)或poly(dA)的背景下对AID和AGCT底物进行了分子动力学模拟。他们发现,在T底物中观察到的相互作用在A底物中大大减弱,表现出一种截然不同的结合模式(图2)。模拟出来的结合与AID在不同底物上的脱氨活性一致,因为在T底物上的活性高于A底物。因此,他们认为AID与ssDNA骨架的静电相互作用决定了脱氨的优先顺序,而ssDNA碱基序列可能会间接影响结合。
图2 脱氨优先顺序与AID-ssDNA相互作用有关