提到寡核苷酸的spacer修饰,很多小伙伴可能比较陌生,它们不像荧光、硫代、biotin修饰等赋予寡核苷酸显而易见的特点;但是在许多生物、化学应用中,使用合适的spacer,会帮助我们收获更漂亮的结果。本文将给大家分享常见的spacer种类与应用实例,让我们共同学习、探讨spacer的使用。
什么是spacer
Spacer指的是间臂类修饰,大部分spacer由直碳链或乙二醇组成。将spacer引入到寡核苷酸中,通常是为了在寡核苷酸之间或寡核苷酸与其他官能团之间建立一段距离,以避免空间位阻、减少基团之间不利的相互作用、增加柔性等;或者在不需要寡核苷酸做延伸的情况中,spacer则作为封闭基团使用。不同spacer的原子数量不同,可以通过调节所插入spacer的数量与种类达到需要的空间距离。常见的spacer有疏水性的Spacer C3、C6、C12,亲水性的Spacer 9、Spacer 18,以及特殊的dSpacer、PC linker。
Spacer C3应用实例:
1)Spacer C3可用于模仿核糖的3'和5'羟基间的三碳间隔, 因此可用来“替代”一段序列中未知的碱基,该应用多见于蛋白与核酸相互作用的研究。
有研究在探索HIV Tat蛋白与RNA相互作用时,将RNA序列中某些核苷酸位点替代为Spacer C3,从而判断该核苷酸位点在Tat蛋白识别RNA中是提供结合位点或是作为间隔[1]。
2)Spacer C3经常被放在寡核苷酸的3'端,作为寡核苷酸延伸的封闭基团,同时Spacer C3对于核酸外切酶有一定的抗性。
经典的例子可见于TaqMan qPCR。TaqMan探针通常将荧光基团放在探针5'端,淬灭基团放在探针3'端,但是在TaqMan探针较长时,为了达到较好的淬灭效果,可通过dT将淬灭基团引入到探针中间;TaqMan探针的3'端暴露出来,探针可能作为引物在模板上进行非预期的扩增,此时通常会在探针3'端修饰Spacer C3以封闭探针扩增。类似的,在许多测序引物中也会引入Spacer C3起到封闭作用。
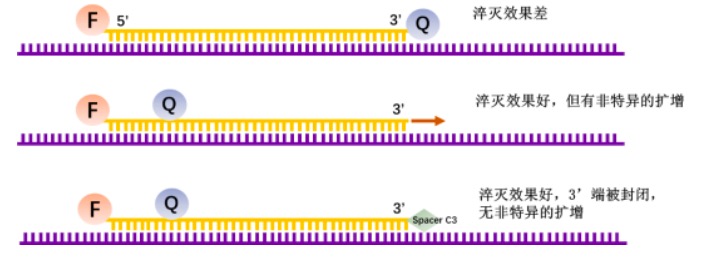
图注:F为荧光基团,Q为淬灭基团
3)Spacer C3也可引入到固相载体与寡核苷酸之间,从而减少两者间的空间位阻,有利于寡核苷酸与目标片段的杂交。
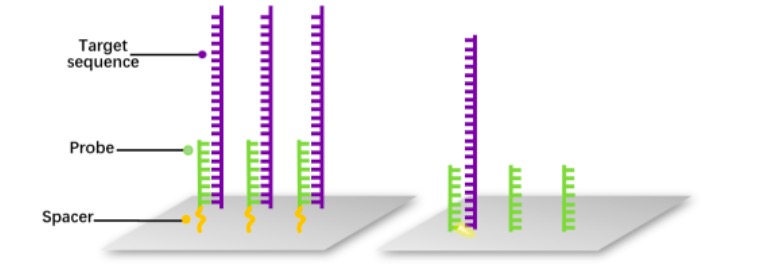
Spacer C6应用实例:
Spacer C6可引入到固相载体与寡核苷酸之间,从而减少两者间的空间位阻,有利于寡核苷酸与目标片段的杂交。
Spacer C12应用实例:
Spacer C12可引入到固相载体与寡核苷酸之间,从而减少两者间的空间位阻,有利于寡核苷酸与目标片段的杂交。
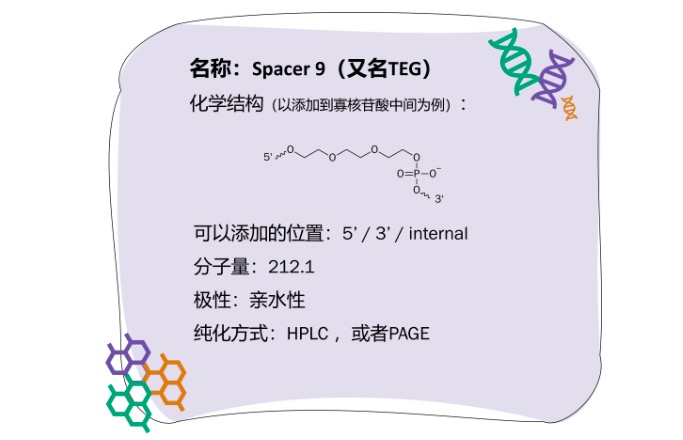
Spacer 9应用实例:
Spacer 9常用于桥连两个结构域,赋予两个结构域之间较大的灵活性,有利于结构域发挥作用。比如在peptide nucleic acid(PNA,肽核酸,一种以多肽骨架取代糖磷酸主链的DNA类似物,属于人工产物,主要应用于DNA分子的识别与操纵,也有应用于反义核酸药物研究)领域,有研究在PNA与donor DNA之间使用多个Spacer 9做连接。在药物研发中,也有Spacer 9连接寡核苷酸与小分子抗原表位的案例[2]。
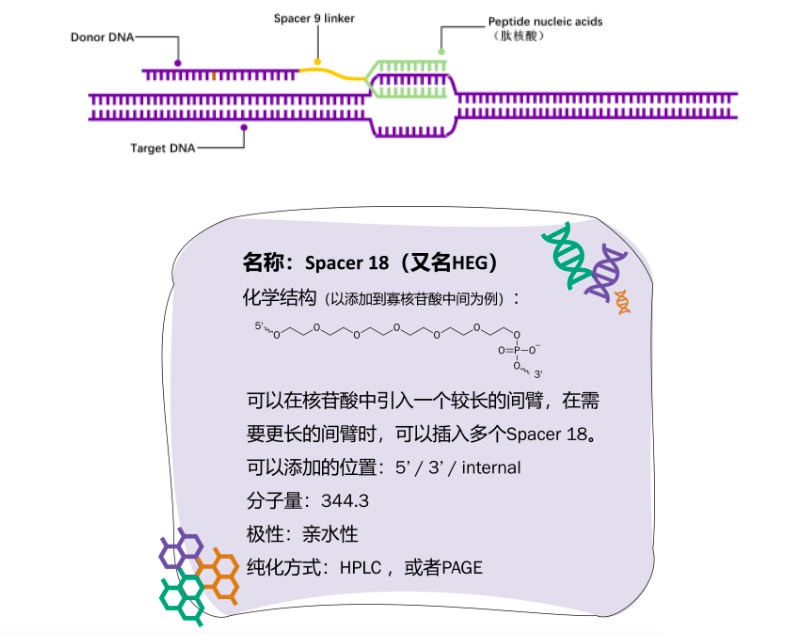
Spacer 18应用实例:
1)Spacer 18有较好的柔性,常用于辅助核酸链形成发卡结构。在早期已有研究表明,在两段互补寡核苷酸链中引入Spacer 18辅助形成的发卡结构,比引入4个T形成的发卡结构更加稳定[3]。
2)Spacer 18作为非核酸的柔性linker替代一段核酸序列,提高寡核酸各部分间的灵活性,同时Spacer 18不会与体系中其他核酸链产生互补配对。经典的例子就是蝎形探针。
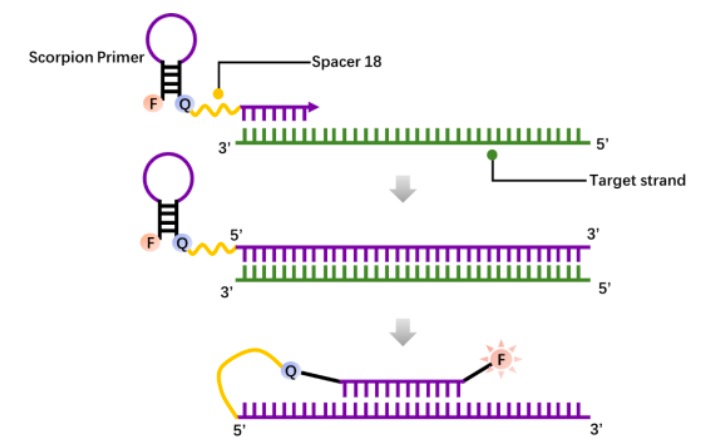
图注:F为荧光基团,Q为淬灭基团
3)Spacer 18常用于引入一个疏水基团,隔开不同的基团以减少不利的位阻或相互作用[4]。类似的,Spacer 18也会添加到固相载体与寡核苷酸之间以减少两者间的空间位阻。
例如在PROER-seq技术中(通过测序在转录组尺度上揭示蛋白相互作用)[4],作者以4个Spacer 18为间臂,再利用点击化学技术,在寡核苷酸上添加一个puromycin(嘌呤霉素)。
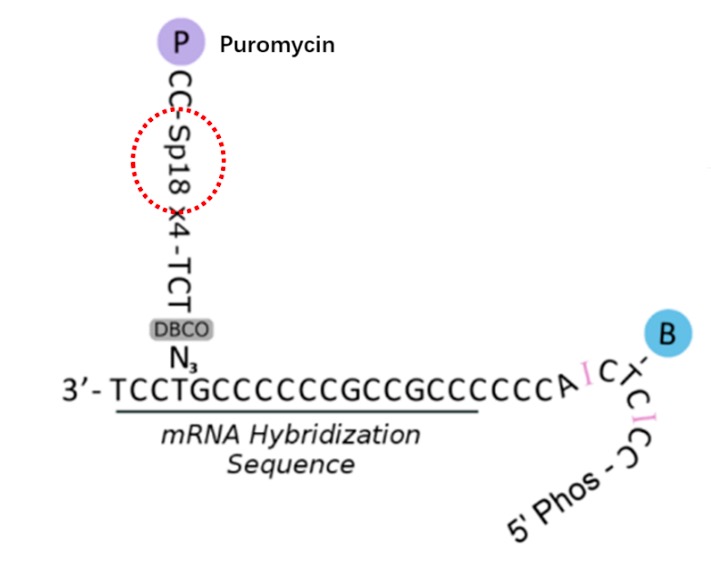
图片来源:《Revealing protein-protein interactions at the transcriptome scale by sequencing》
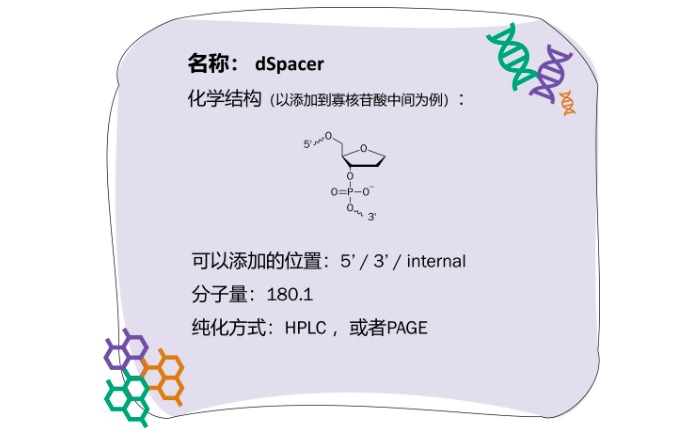
dSpacer应用实例:
1)在前文我们提到Spacer C3可用于 “替代”一个序列中未知的碱基。但是烷基的柔性可能使得糖-磷酸骨架扭曲,使用dSpacer则可以缓解这种情况。从dSpacer的化学结构我们也可以发现,将dSpacer添加到寡核苷酸中,可以引入一个稳定的无碱基位点,模拟体内脱嘌呤导致的基因突变,因此dSpacer经常见于DNA损伤与修复的研究[5]。
2)有研究利用dSpacer模拟无碱基位点的特性,将dSpacer添加到siRNA中以降低siRNA的脱靶效应。siRNA是敲低基因的常用手段,但siRNA可能作为miRNA起作用,从而导致miRNA-like的脱靶。研究发现用dSpacer取代siRNA序列的pivot位置(position 6,miRNA与靶标相互作用的关键位点),可降低siRNA的miRNA-like脱靶效应,而保留80%~100%的on-target基因沉默能力[6]。
PC linker应用实例:
PC linker现广泛应用于分子检测。在DNA/RNA的分析中,会给杂交探针添加荧光、生物素、酶等去标记特定的DNA/RNA或者作为测序引物。寡核苷酸共价标记的缺点之一是共价键不能被选择性切除,因此标记不能被去除或核酸不能被释放。采用化学方法裂解可能对样品造成影响,且会有化学试剂残留。PC linker较好地解决了上述难题。
经典的应用可以联想到近几年大热的空间转录组技术。比如CosMx SMI,该技术用于构建单细胞与亚细胞分辨率的3D RNA图谱,通过使用荧光分子barcode对生物样本的核酸进行多轮杂交,实现生物样本上亚细胞级分辨率的RNA原位检测。如示意图所示,通过在荧光探针上引入PC site(即PC linker),新一轮杂交前通过光照射去除上一轮的荧光探针,在不损坏样品的前提下,保证每轮杂交的低背景。
看完这些实例,小编感觉寡核苷酸世界丰富多彩,我们可以通过修饰基团赋予寡核苷酸很多功能,但没有spacer,这些修饰基团也难以发挥最大的作用。当然,spacer的作用远不止上述的内容,本文仅是抛砖引玉,更多的应用等待大家发掘,欢迎大家一起探讨。
参考文献:
[1] Pritchard, C. E., Grasby, J. A., Hamy, F., Zacharek, A. M., Singh, M., Karn, J., & Gait, M. J. (1994). Methylphosphonate mapping of phosphate contacts critical for RNA recognition by the human immunodeficiency virus tat and rev proteins. Nucleic acids research, 22(13), 2592–2600. https://doi.org/10.1093/nar/22.13.2592
[2] Palma, E., Klapper, D. G., & Cho, M. J. (2005). Antibodies as drug carriers III: design of oligonucleotides with enhanced binding affinity for immunoglobulin G. Pharmaceutical research, 22(1), 122–127. https://doi.org/10.1007/s11095-004-9017-z
[3] Salunkhe, M., Wu, T., & Letsinger, R. L. (1992). Control of folding and binding of oligonucleotides by use of a nonnucleotide linker. Journal of the American Chemical Society, 114(23), 8768-8772. https://pubs.acs.org/doi/10.1021/ja00049a003
[4] Johnson, K. L., Qi, Z., Yan, Z., Wen, X., Nguyen, T. C., Zaleta-Rivera, K., Chen, C. J., Fan, X., Sriram, K., Wan, X., Chen, Z. B., & Zhong, S. (2021). Revealing protein-protein interactions at the transcriptome scale by sequencing. Molecular cell, 81(19), 4091–4103.e9. https://doi.org/10.1016/j.molcel.2021.07.006
[5] Cheng, K., Xu, Y., Chen, X., Lu, H., He, Y., Wang, L., & Hua, Y. (2020). Participation of RecJ in the base excision repair pathway of Deinococcus radiodurans. Nucleic acids research, 48(17), 9859–9871. https://doi.org/10.1093/nar/gkaa714
[6] Lee, H. S., Seok, H., Lee, D. H., Ham, J., Lee, W., Youm, E. M., Yoo, J. S., Lee, Y. S., Jang, E. S., & Chi, S. W. (2015). Abasic pivot substitution harnesses target specificity of RNA interference. Nature communications, 6, 10154. https://doi.org/10.1038/ncomms10154
[7] He, S., Bhatt, R., Brown, C., Brown, E. A., Buhr, D. L., Chantranuvatana, K., Danaher, P., Dunaway, D., Garrison, R. G., Geiss, G., Gregory, M. T., Hoang, M. L., Khafizov, R., Killingbeck, E. E., Kim, D., Kim, T. K., Kim, Y., Klock, A., Korukonda, M., Kutchma, A., … Beechem, J. M. (2022). High-plex imaging of RNA and proteins at subcellular resolution in fixed tissue by spatial molecular imaging. Nature biotechnology, 10.1038/s41587-022-01483-z. Advance online publication. https://doi.org/10.1038/s41587-022-01483-z
(15):1408-1421.
3. Fava A, Buyon J, Mohan C, Zhang T, et al. Integrated urine proteomics and renal single-cell genomics identify an IFN-γ response gradient in lupus nephritis. JCI Insight. 2020 Jun 18;5(12):e138345.

